comme vous pouvez le voir sur la Figure1, les nucléotides ne varient que légèrement, et seulement dans la base azotée. Dans le cas de L’ADN, ces bases sont l’adénine, la guanine, la cytosine et la thymine. Notez la similitude des formes de l’adénine et de la guanine, ainsi que la similitude entre la cytosine et la thymine. A et G sont classés comme purines, tandis que C et T sont classés comme pyrimidines. Tant que nous nommons les choses, notez « désoxyribose” et « ribose”. Comme son nom l’indique, le désoxyribose n’est qu’un ribose sans oxygène., Plus précisément, lorsqu’il y a un groupe hydroxyle attaché au carbone 2 du ribose, il n’y a qu’un hydrogène attaché au carbone 2 du désoxyribose. C’est la seule différence entre les deux sucres.
dans la construction aléatoire d’un seul brin d’acide nucléique in vitro, il n’y a pas de règles particulières concernant l’ordre des nucléotides par rapport à leurs bases. L’identité de leurs bases azotées n’est pas pertinente car les nucléotides sont attachés par des liaisons phosphodiester à travers le groupe phosphate et le pentose., Il est donc souvent appelé l’épine dorsale sucre-phosphate. Si nous décomposons le mot « phosphodiester », nous voyons qu’il décrit assez facilement la connexion: les sucres sont reliés par deux liaisons ester ( —O—) avec un phosphore entre les deux. L’une des idées qui confond souvent les étudiants est la directionnalité de cette liaison, et donc des acides nucléiques en général. Par exemple, lorsque nous parlons D’ADN polymérase, l’enzyme qui catalyse l’addition de nucléotides dans les cellules vivantes, nous disons qu’elle fonctionne dans une direction 5-prime (5’) à 3-prime (3’)., Cela peut sembler arcanes moléculaire-biologiste-parler, mais il est en fait très simple. Regardez deux des nucléotides réunis par la liaison phosphodiester (Figure \(\PageIndex{1}\), en bas à gauche). Un nucléotide adénine est joint à un nucléotide cytosine. La liaison phosphodiester reliera toujours le carbone 5 d’un désoxyribose (ou ribose dans L’ARN) au carbone 3 du sucre suivant. Cela signifie également qu’à une extrémité d’une chaîne de nucléotides liés, il y aura un groupe phosphate 5’ libre (-PO4), et à l’autre extrémité, un hydroxyle 3’ libre (-OH)., Ceux-ci définissent la directionnalité d’un brin d’ADN ou D’ARN.

L’ADN se trouve normalement sous forme de molécule double brin dans la cellule alors que L’ARN est principalement monocaténaire., Il est important de comprendre cependant que, dans les conditions appropriées, L’ADN pourrait être simple brin, et L’ARN peut être double brin. En fait, les molécules sont si similaires qu’il est même possible de créer des molécules hybrides double brin avec un brin d’ADN et un brin d’ARN. Fait intéressant, les doubles hélices ARN-ARN et les doubles hélices ARN-ADN sont en fait légèrement plus stables que la double hélice ADN-ADN plus conventionnelle.,
La base de la nature double brin de L’ADN, et en fait la base des acides nucléiques en tant que milieu de stockage et de transfert de l’information génétique, est l’appariement des bases. L’appariement des bases fait référence à la formation de liaisons hydrogène entre les adénines et les thymines, et entre les guanines et les cytosines. Ces paires sont significativement plus stables que toute association formée avec les autres bases possibles. De plus, lorsque ces associations de paires de bases se forment dans le contexte de deux brins d’acides nucléiques, leur espacement est également uniforme et très stable., Vous vous souvenez peut-être que les liaisons hydrogène sont des liaisons relativement faibles. Cependant, dans le contexte de L’ADN, la liaison hydrogène est ce qui rend L’ADN extrêmement stable et donc bien adapté comme support de stockage à long terme pour l’information génétique. Puisque même chez les procaryotes simples, les doubles hélices D’ADN ont au moins des milliers de nucléotides, cela signifie qu’il existe plusieurs milliers de liaisons hydrogène maintenant les deux brins ensemble., Bien que toute interaction de liaison hydrogène nucléotide-nucléotide individuelle puisse facilement être temporairement perturbée par une légère augmentation de la température ou un changement minime de la force ionique de la solution, une double hélice complète d’ADN nécessite des températures très élevées (généralement supérieures à 90oC) pour dénaturer complètement la double hélice en brins individuels.
parce qu’il y a un appariement exact un à un des nucléotides, il s’avère que les deux brins sont essentiellement des copies de sauvegarde l’un de l’autre-un filet de sécurité dans le cas où des nucléotides sont perdus d’un brin., En fait, même si des parties des deux brins sont endommagées, tant que l’autre brin est intact dans la zone endommagée, alors les informations essentielles sont toujours là dans la séquence complémentaire du brin opposé et peuvent être écrites en place. Gardez à l’esprit cependant, que si un brin D’ADN peut ainsi agir comme une « sauvegarde” de l’autre, les deux brins ne sont pas identiques – ils sont complémentaires. Une conséquence intéressante de ce système de brins complémentaires et antiparallèles est que les deux brins peuvent chacun porter des informations uniques.,
les paires de gènes bidirectionnels sont deux gènes sur des brins d’ADN opposés, mais partageant un promoteur, qui se trouve entre eux. Puisque L’ADN ne peut être fabriqué que dans une direction, 5’ à 3’, ce promoteur bidirectionnel, souvent un îlot CpG (voir chapitre suivant), envoie ainsi l’ARN polymérase pour chaque gène dans des directions physiques opposées. Ceci a été démontré pour un certain nombre de gènes impliqués dans les cancers (sein, ovaire), et est un mécanisme de coordination de l’expression de réseaux de produits génétiques.
Les brins d’une double hélice d’ADN sont antiparallèles., Cela signifie que si nous examinions une double hélice D’ADN de gauche à droite, un brin serait construit dans la direction 5’ à 3’, tandis que le brin complémentaire est construit dans la direction 3’ à 5’. Ceci est important pour la fonction des enzymes qui créent et réparent L’ADN, comme nous en discuterons bientôt. Dans la Figure \(\PageIndex{1}\), le brin gauche est de 5’ à 3’ de haut en bas, et l’autre est de 5’ à 3’ de bas en haut.
d’un point de vue physique, les molécules D’ADN sont chargées négativement (tous ces phosphates), et normalement une double hélice avec une torsion droitière., Dans cet état normal (également appelé conformation « B »), une torsion complète de la molécule englobe 11 paires de bases, avec 0,34 nm entre chaque base nucléotidique. Chacune des bases azotées est plane et, lorsqu’elle est associée à la base complémentaire, elle forme un « échelon” at planaire sur l’ « échelle” de L’ADN. Ces sont perpendiculaires à l’axe longitudinal de l’ADN. La plus grande partie de L’ADN flottant dans une cellule, et la plus grande partie de l’ADN dans toute solution aqueuse d’osmolarité et de pH quasi physiologiques, se trouve dans cette conformation B., Cependant, d’autres conformations ont été trouvées, généralement dans des circonstances environnementales très spécifiques. Une conformation comprimée, L’ADN-a, a été observée comme artefact de cristallisation in vitro, avec un peu plus de bases par tour, une longueur de tour plus courte et des paires de bases qui ne sont pas perpendiculaires à l’axe longitudinal. Un autre, L’ADN-Z, semble se former de manière transitoire dans des étendues d’ADN riches en GC dans lesquelles, fait intéressant, l’ADN se tord dans la direction opposée.
Il a été suggéré que les Formes A et Z de L’ADN sont, en fait, physiologiquement pertinentes., Il existe des preuves suggérant que la forme A peut se produire dans les doubles hélices hybrides ARN-ADN ainsi que lorsque L’ADN est complexé à certaines enzymes. La conformation Z peut se produire en réponse à la méthylation de l’ADN. En outre, la conformation « normale” de L’ADN-B est une structure idéalisée basée sur une hydratation complète, comme cela est certainement très probable à l’intérieur d’une cellule. Cependant, cet état d’hydratation change constamment, quoique de manière minutieuse, de sorte que la conformation de l’ADN varie souvent légèrement par rapport aux paramètres de conformation B de la Figure \(\PageIndex{2}\).,
chez les procaryotes, l’ADN se trouve dans le cytoplasme (assez évident car il n’y a pas d’autre choix chez ces organismes simples), tandis que chez les eucaryotes, l’ADN se trouve à l’intérieur du noyau. Malgré les différences dans leurs emplacements, le niveau de protection contre les forces externes, et surtout, leurs tailles, l’ADN procaryote et eucaryote est emballé avec des protéines qui aident à organiser et à stabiliser la structure chromosomique globale., On comprend relativement peu de choses en ce qui concerne l’emballage chromosomique procaryote, bien qu’il existe des similitudes structurelles entre certaines des protéines présentes dans les chromosomes procaryotes et eucaryotes. Par conséquent, la plupart des cours d’introduction à la biologie cellulaire s’en tiennent à l’emballage chromosomique eucaryote.

L’ADN nu, procaryote ou eucaryote, est un brin de matière extrêmement mince, d’environ 11 nm de diamètre. Cependant, étant donné la taille des génomes eucaryotes, si l’ADN était stocké de cette façon à l’intérieur du noyau, il deviendrait ingérable emmêlé. Imaginez un seau dans lequel vous avez jeté une centaine de mètres de fil sans aucune tentative de l’organiser en l’enroulant ou en le groupant., Maintenant, considérez si vous seriez en mesure d’atteindre dans ce seau tirer sur un brin, et attendez-vous à tirer un seul brin, ou si au lieu de cela vous êtes susceptible de tirer au moins un petit enchevêtrement de fil. La cellule fait essentiellement ce que vous feriez avec le fil pour le garder organisé: il est emballé soigneusement en écheveaux plus petits et gérables. Dans le cas de L’ADN, chaque chromosome est bouclé autour d’un complexe histone pour former le premier ordre d’organisation chromosomique: le nucléosome.
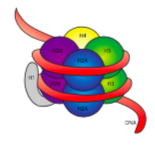
la fibre de 30 nm est maintenue ensemble par deux ensembles d’interactions. Tout d’abord, L’histone de l’éditeur de liens, H1, rassemble les nucléosomes dans une structure d’environ 30 nm., Cette structure est ensuite stabilisée par des liaisons disulfures qui se forment entre L’histone H2A d’un nucléosome et L’histone H4 de son voisin.
les Histones sont une famille de protéines basiques (chargées positivement). Ils fonctionnent tous principalement dans l’organisation de l’ADN, et le nucléosome se forme lorsque L’ADN s’enroule (un peu plus de 2 fois) autour d’un noyau de huit histones – deux chacune de H2A, H2B, H3 et H4. Le nombre et la position des charges positives (principalement des lysines et des arginines) sont cruciaux pour leur capacité à lier étroitement L’ADN, qui, comme indiqué précédemment, est très chargé négativement., Que l” idée « opposés attirent » n » est pas seulement sortir ensemble astuce des colonnes de conseils.

en examinant la structure 3D du complexe de noyau d’histone, nous voyons que bien que des domaines d’interaction protéiques relativement non chargés maintiennent les histones ensemble au centre, les résidus chargés positivement se trouvent autour de l’extérieur du complexe, disponibles pour interagir avec les phosphates chargés négativement de L’ADN.,
dans un chapitre ultérieur, nous discuterons de la façon dont les enzymes lisent l’ADN pour transcrire ses informations sur des morceaux d’ARN plus petits et plus faciles à gérer. Pour l’instant, nous devons seulement être conscients qu’à un moment donné, une grande partie de l’ADN est emballée étroitement, alors que certaines parties de l’ADN ne le sont pas. Étant donné que les pièces disponibles peuvent varier en fonction de ce qui se passe dans/dans la cellule à un moment donné, l’emballage de l’ADN doit être dynamique. Il doit y avoir un mécanisme pour desserrer rapidement la liaison de L’ADN aux histones lorsque cet ADN est nécessaire à l’expression des gènes, et pour resserrer la liaison quand ce n’est pas le cas., Il s’avère que ce processus implique l’acétylation et la désacétylation des histones.

les histones Acétyltransférases (HATs) sont des enzymes qui placent un groupe acétyle sur une lysine d’une protéine histone. Les groupes acétyle sont chargés négativement et l’acétylation ajoute non seulement un groupe chargé négativement, mais supprime également la charge positive de la lysine. Cela a pour effet non seulement de neutraliser un point d’attraction entre la protéine et L’ADN, mais même de le repousser légèrement (avec des charges similaires)., De l’autre côté du mécanisme, les histones Deactylases (HDACs) sont des enzymes qui éliminent l’acétylation et rétablissent ainsi l’interaction entre la protéine histone et L’ADN. Comme ce sont des enzymes si importantes, il va de soi qu’elles ne sont pas autorisées à fonctionner bon gré mal gré sur n’importe quelle histone disponible, et en fait, elles se trouvent souvent dans un complexe avec d’autres protéines qui contrôlent et coordonnent leur activation avec d’autres processus tels que l’activation de la transcription.
