Introduction
Les télomères sont des structures nucléoprotéiques aux extrémités des chromosomes eucaryotes linéaires. Ils résolvent deux problèmes finaux majeurs en même temps. Le premier concerne la protection des extrémités chromosomiques. On estime que les cellules humaines normales doivent réparer au moins 50 cassures endogènes à double brin (DSB) par cellule et par cycle cellulaire (Vilenchik et Knudson, 2003)., Les télomères distinguent les extrémités chromosomiques naturelles des DSB nuisibles et empêchent leur réparation ectopique, par exemple par des fusions de bout en bout de chromosomes (vanSteensel et deLange, 1997). Le second est le problème de la réplication finale qui traite de la maintenance des longueurs de télomères appropriées. Cela a été reconnu indépendamment par deux chercheurs (Watson, 1972; Olovnikov, 1973). Étant donné que les ADN polymérases dépendantes de l’ADN réplicative ne peuvent pas compléter la synthèse de l’ADN aux extrémités des chromosomes, la compensation de la perte de séquence des télomères réplicative doit provenir d’une ADN polymérase dépendante de l’ARN., Cette enzyme, appelée télomérase, ainsi que la première séquence de minisatellites télomères, ont été découvertes dans la Tétrahymène ciliée (Blackburn et Gall, 1978; Greider et Blackburn, 1985). Cependant, ce n’est qu’un aspect de la longueur des télomères de maintenance. La régulation épigénétique de l’homéostasie de la longueur des télomères, y compris l’interaction de longues répétitions télomériques non codantes contenant de l’ARN et des voies d’activité exonucléase, a également été largement étudiée en raison de son potentiel thérapeutique (Wellinger et al., 1996; Polotnianka et coll., 1998; Pfeiffer et Lingner, 2012).,
la télomérase, l’enzyme chargée d’ajouter des séquences de répétition des télomères à l’extrémité 3′ des télomères, est une enzyme complexe conservée avec de nombreux composants . En principe, seuls deux composants principaux sont essentiels à l’activité enzymatique de la télomérase, un composant protéique catalytiquement actif, appelé transcriptase inverse des télomères (TERT), et un composant modèle, formé par la sous-unité d’ARN de la télomérase (TR). Alors que TERT est évolutivement assez bien conservé, TR est très variable,avec des longueurs allant de ca. 150 nt (Tetrahymena) à plus de 2 000 NT (champignons du genre Neurospora)., Seule une courte région de la molécule TR entière Sert de modèle pour L’ADN télomère nouvellement synthétisé (Greider et Blackburn, 1985; Qi et al., 2013). Cette région dans TR est généralement formée par un motif télomère complet suivi d’un motif partiel, ce dernier servant de région de recuit pour L’ADN télomère existant. Bien que, en principe, un seul nucléotide supplémentaire soit nécessaire (en tant que motif partiel), on en trouve généralement plus d’un. Par exemple, deux nucléotides supplémentaires forment le motif de recuit chez la souris ou cinq chez l’homme (Blasco et al., 1995; Feng et coll., 1995)., Chez les plantes, cependant, la taille de la région du gabarit est variable, par exemple, deux chez Arabidopsis thaliana, sept chez Arabis sp. ou six à Nicotiana (Fajkus et al., 2019). Les autres régions TR ont des fonctions structurales, régulatrices et interactives sur les protéines . Voir aussi une représentation schématique de la télomérase et de son cycle d’activité à la Figure 1.

Figure 1 Représentation schématique du cycle d’activité de la télomérase avec le modèle de télomère de type Arabidopsis. TERT, transcriptase inverse des télomères; TR, sous-unité d’ARN télomérase., Chiffre basé sur Sekhri (2014).
Quelle est la Variable des séquences de télomères?
Les séquences de télomères sont généralement de courts minisatellites disposés en tandem, suivant généralement la formule (TxAyGz)N. L’arrangement de minisatellites provient de la façon dont la télomérase synthétise l’ADN, en bref, et la plupart du temps des motifs identiques, un par un. , Plusieurs hypothèses considèrent qu’un tel arrangement est important car il favorise la reconnaissance des protéines spécifiques des télomères par les homo – et hétérodimères et pour le potentiel de former des G-quadruplexes qui peuvent stabiliser les extrémités des chromosomes ou servir de substrats pour les protéines spécifiques des télomères (Spiegel et al., 2020; Tran et coll., 2013). Les séquences de télomères sont bien conservées au cours de l’évolution, et de grands groupes d’organismes utilisent le motif des télomères typique du groupe pour construire leur ADN télomère., Un nombre croissant d’études et de grands dépistages ont montré que tous les vertébrés testés et de nombreux métazoaires basaux utilisent TTAGGG (Meyne et al., 1989; Traut et coll., 2007) tandis que les Euarthropodes (arthropodes), y compris les Hexapodes (insectes), ont TTAGG (Frydrychova et al., 2004; Vitkova et coll., 2005). Régulièrement, de nombreuses exceptions s’accumulent au fil du temps, par exemple (A(G)1-8) chez Dictyostelium (Emery et Weiner, 1981), TTAGGC chez Ascaris lumbricoides (Nematoda) (Muller et al., 1991), TCAGG chez les coléoptères (coléoptères) (Mravinac et al., 2011), TAGGG / TAAGG / TAAGGG in Giardia (diplomonads) (Uzlikova et al.,, 2017), ou TTNNNNAGGG dans le clade Yarrowia (levures) (Cervenak et al., 2019). De plus, les systèmes indépendants de la télomérase, dans lesquels la séquence des télomères minisatellites a été perdue et remplacée par des répétitions complexes, sont représentés, par exemple, par des diptères et des Chironomidae (examinés dans (Mason et al., 2016)). Pour un examen général sur la séquence des télomères eucaryotes, voir (Fajkus et al., 2005; Fulneckova et coll., 2013).
la composition des télomères chez les plantes est encore plus diversifiée., Ici, nous utilisons le terme « plantes » dans un sens large, également connu sous le nom Archaeplastida ou Royaume Plantae sensu lato, et comprenant Rhodophyta (algues rouges), Glaucophyta, le grade d’algues Chlorophytes et le grade d’algues Streptophytes (tout à fait connu sous le nom d’algues vertes), et Embryophyta (plantes terrestres) (One Thousand Plant Transcriptomes Initiative, 2019). La séquence végétale typique des télomères est TTTAGGG, également appelée Type Arabidopsis (ou simplement, type de plante) car elle a été découverte chez Arabidopsis thaliana (Richards et Ausubel, 1988) et maintenant chez de nombreuses autres espèces dans presque tous les ordres de plantes., Bien que TTTAGGG soit toujours le plus fréquent, il existe une variabilité significative des séquences de télomères dans les lignées d’algues rouges et vertes. En ce qui concerne les algues rouges (Rhodophyta), les informations sur la séquence des télomères sont pour la plupart manquantes ou fragmentaires, bien que certains candidats télomères aient été découverts dans silico, comme AATGGGGGG pour Cyanidioschyzon merolae (Nozaki et al., 2007), TTATT(T)AGGG pour Galdieria sulphuraria (Fulneckova et al., 2013); TTAGGG a été trouvé dans les lectures génomiques de Porphyra umbilicalis (Fulneckova et al., 2013), mais plus de preuves sont nécessaires pour confirmer leur position terminale sur les chromosomes., La diversité des télomères chez les algues vertes reflète à la fois les changements dynamiques et son caractère paraphylétique. Bien que TTTAGGG prévaut chez les Chlorophyta, comme dans les genres Ostreococcus (Derelle et al., 2006) et la Chlorella (Higashiyama et coll., 1995), de nombreux autres motifs divergents y ont également été détectés, tels que TTAGGG dans le genre Dunaliella et Stephanosphaeria (Fulneckova et al., 2012), et TTTTAGGG dans Chlamydomonas (Petracek et al., 1990). Chez les Streptophytes basaux (Klebsormidiophyceae), des changements progressifs dans les motifs de TTTAGGG à TTTTAGGG et TTTTAGG ont été décrits., La présence de TTAGGG chez Rhodophyta et Glaucophyta conduit à l’hypothèse qu’il s’agit du motif ancestral chez les plantes (Archaeplastida) (Fulneckova et al., 2013).
en ce qui concerne les plantes terrestres, l’une des premières analyses effectuées a montré que la séquence de type Arabidopsis était la plus courante et était principalement conservée par leur phylogénie (Cox et al., 1993; Fuchs et coll., 1995), bien que certains de ces auteurs aient déjà détecté plusieurs exceptions dans la famille des Amaryllidaceae (anciennement Alliaceae), dans laquelle la séquence de type Arabidopsis était absente chez plusieurs espèces., Plus tard, la première séquence de télomères inhabituelle pour les plantes terrestres, le ttaggg de type vertébré, a été caractérisée dans L’aloès et dans quelques autres Asparagales (Weiss et Scherthan, 2002; Puizina et al., 2003; Sykorova et coll., 2003c). Une hypothèse sur les pertes et les recouvrements répétés de la séquence des télomères tttaggg et TTAGGG chez les Asparagales a été formulée (Adams et al., 2001). Avec le post-affinement de L’ordre des Asparagales dans les APGIII (Angiosperm Phylogeny Group 2009) (Bremer et al.,, 2009), il a été démontré que seulement deux changements évolutifs majeurs dans la composition de la séquence des télomères se sont produits (plutôt que plusieurs pertes et gains répétés), dans l’ordre suivant: le premier dans la famille Iridaceae, dans lequel un passage du type végétal TTTAGGG au type vertébré ttaggg s’est produit, suivi par les familles Xeronemataceae, Asphodelaceae et,); et la seconde au sein de la sous-famille Allioideae (anciennement traitée comme une famille distincte, Alliaceae) dans laquelle une toute nouvelle séquence de télomères a émergé, CTCGGTTATGGG (Fajkus et al., 2016). En dehors des Asparagales, de nouvelles séquences de télomères ont également été détectées dans des groupes de plantes terrestres aussi disparates que (I) Solanaceae, dans lesquels la séquence de télomères de Cestrum elegans TTTTTTAGGG a été décrite (Sykorova et al., 2003a; Sykorova et coll., 2003b; Peska et coll., 2008; Peska et coll.,, 2015) et (ii) Lentibulariaceae, où le genre Genlisea a montré une diversité remarquable avec certaines espèces caractérisées par les répétitions de télomères de type Arabidopsis tandis que d’autres présentaient des variantes de séquence entremêlées TTCAGG et TTTCAGG (Tran et al., 2015).
malgré toutes les exceptions de motifs télomères détectées, la diversité réelle des séquences télomériques chez les plantes terrestres est probablement grandement sous-estimée. Une publication récente (Vitales et coll.,, 2017), dans laquelle un criblage des séquences de télomères terrestres des plantes a été effectué, a révélé que les séquences de télomères n’étaient connues clairement que pour moins de 10% des espèces et 40% des genres contenus dans la base de données de l’ADNR des plantes (www.plantrdnadatabase.com), une ressource fournissant des informations de cytogénétique moléculaire sur les plantes terrestres (Garcia et al., 2012). Un résumé de la séquence de télomère de distribution dans les plantes, à la suite de l’APG IV (La Phylogénie des Angiospermes Groupe, 2016) (Byng et coll., 2016), ainsi que la plus récente phylogénie des plantes (One Thousand Plant Transcriptomes Initiative, 2019) se trouve à la Figure 2.,
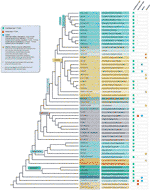
figure 2 motifs de télomères chez Archaeplastida (Plantes au sens large), basés sur L’APG IV (The Angiosperm Phylogeny Group 2016) et sur L’initiative One Thousand Plant Transcriptomes (2019). Les longueurs de branche n’expriment pas les échelles en temps réel., Par souci de simplicité et pour économiser de l’Espace, certains « groupes” polyphylétiques (grades) marqués d’un astérisque dans l’arbre ont été représentés par une seule branche; pour la même raison, plusieurs ordres mineurs (énumérés dans le carré bleu en haut à gauche de la figure) ne sont pas représentés sur l’arbre. La première étiquette de pointe se réfère généralement à des ordres de plantes et dans quelques cas, à des divisions, des grades et même des familles; la deuxième étiquette affiche des familles représentatives et dans quelques cas, des ordres ou des genres représentatifs.
des projections à la découverte: comment identifier les Motifs télomériques?,
la preuve qu’une séquence candidate donnée est une séquence télomérique réelle comprend plusieurs étapes qui déclarent correctement sa localisation à tous les termini chromosomiques, et éventuellement l’implication de la télomérase dans sa synthèse. La cytogénétique moléculaire (principalement par hybridation in situ par Fluorescence, FISH) est devenue importante pour visualiser la localisation terminale de sondes marquées de séquences candidates à tous les termini chromosomiques. Cependant, poisson autonome il ne suffit pas de prouver la position très terminale., Par exemple, AcepSAT356 a été proposé dans onion comme candidat télomère, d’après les résultats de L’analyse des poissons (Pich et Schubert, 1998). Néanmoins, son emplacement terminal apparent in situ n’a jamais été lié de manière convaincante à la fonction télomère. En fait, la découverte de la séquence des télomères minisatellites D’Allium CTCGGTTATGGG et de la télomérase signifierait que AcepSAT356 est subterminal (Fajkus et al., 2019)., Les signaux télomériques de poisson positifs peuvent également masquer de minuscules changements dans les motifs des télomères tels que les polymorphismes d’un seul nucléotide, ou des résultats faussement négatifs peuvent résulter du fait que les télomères courts sont inférieurs à la limite de détection de la technique.
Il existe deux approches supplémentaires qui déterminent la position terminale à une résolution supérieure à celle de FISH; celles-ci sont basées sur l’activité de l’exonucléase BAL31., La première est l’analyse Classique des fragments de Restriction terminale (TRF), dans laquelle les échantillons traités par BAL31 montrent un raccourcissement progressif des fragments terminaux et une diminution de l’intensité du signal avec l’augmentation du temps de traitement par exonucléase. L’analyse ultérieure des longueurs des fragments est réalisée par hybridation Southern-blot (Fojtova et al., 2015). La seconde est l’écrémage comparatif du génome (données NGS) de L’ADN génomique non digéré et digéré par BAL31, en parallèle., Dans L’ensemble de données traité BAL31, il existe une sous-représentation significative des séquences de télomères, par conséquent les séquences terminales sont identifiées par comparaison avec l’ensemble de données non traité, à l’aide des outils bioinformatiques RepeatExplorer ou Tandem Repeats Finder .
L’autre test important d’une séquence de télomère candidat dans une espèce est la démonstration de l’activité de la télomérase. En cela, une approche expérimentale utile, développée d’abord pour les cellules humaines, est le Telomere Repeat Amplification Protocol (TRAP) (Kim et al., 1994), suivi du séquençage des produits détectés (Peska et al.,, 2015; Fajkus et coll., 2016), qui est un peu moins sensible aux résultats faussement positifs que le poisson. Toutes ces méthodes, y compris les poissons (Fuchs et al., 1995; Shibata et Hizume, 2011) et d’autres comme l’hybridation slot-blot (Sykorova et al., 2003c), et le PIÈGE (Fulneckova et coll., 2012; Fulneckova et coll., 2016), peut être utilisé pour dépister les télomères dans de larges groupes d’organismes complexes, y compris les plantes., Cependant, seule une combinaison de méthodes convenablement choisies peut conduire de manière convaincante à une conclusion sur la fonction télomère d’une séquence candidate, car les résultats basés sur une seule approche peuvent être trompeurs. Un aperçu plus complet des stratégies d’identification des séquences candidates des télomères de novo, y compris la toute première tentative chez Tetrahymena (Greider et Blackburn, 1985), est résumé dans un article méthodologique, en mettant l’accent sur L’approche NGS utilisée chez les plantes à génomes extrêmement grands (Peska et al., 2017).
y a-t-il Homoplasie dans les séquences de télomères?,
la séquence ancestrale des télomères est considérée comme TTAGGG et est la plus communément trouvée dans l’arbre de vie (Fulneckova et al., 2013). Pourtant, il semble clair que la fréquence de l’homoplasie dans l’évolution du motif des télomères est relativement élevée. Par exemple, des motifs courts et simples comme le tttaggg de type végétal sont apparus indépendamment et à plusieurs reprises dans les cryptomonades, les champignons oomycètes et les alvéolés; de même, le ttaggg de type vertébré a émergé secondairement dans certains groupes de plantes (Asparagales, Rodophyta et algues Chlorophyta) (Sykorova et al., 2003C; Fulneckova et coll.,, 2012; Fulneckova et coll., 2013; Somanathan et Baysdorfer, 2018). La raison pour laquelle certaines séquences de télomères ont émergé plus fréquemment que d’autres séquences, généralement plus complexes, est probablement liée aux pressions de sélection, ce qui favoriserait la précision pour une interaction ADN-protéine spécifique à une séquence particulière (Forstemann et al., 2003). S’il y avait un changement dans chaque motif de télomère, l’interférence dans la structure de nucléoprotéine télomérique conduirait nécessairement à l’instabilité de génome., C’est la raison pour laquelle les séquences de télomères sont si stables sur le plan évolutif, comprenant très peu de séquences nouvelles et réussies, un modèle compatible avec l’idée de pertes répétées et l’émergence des séquences de télomères typiques, comme proposé pour les Asparagales (Adams et al., 2001).
la découverte de l’homoplasie à travers les séquences de télomères soulève la question, Quelles sont les causes moléculaires et les processus qui se déroulent pendant ces changements? Un changement dans la séquence des télomères, même si cela semble trivial dans certains cas (par exemple,, un t supplémentaire), peut causer de graves interférences avec l’intégrité du génome, en raison d’un équilibre perturbé dans les interactions ADN-protéine des télomères. On ne sait pas non plus si un changement dans la séquence des télomères peut avoir un avantage évolutif; à cet égard, (Tran et al., 2015) ont suggéré que l’apparition d’une cytosine « méthylable” dans un brin de télomère riche en g augmenterait la possibilité de régulation par modification épigénétique.
quelles sont les raisons moléculaires des changements dans les Motifs des télomères?,
pour expliquer le changement de séquence des télomères, le premier candidat est la sous-unité modèle de la télomérase, l’ARN télomérase (TR). Le TR précédemment identifié de la levure et des vertébrés appartient à un groupe différent de transcrits, dont la caractéristique de connexion était qu’ils étaient transcrits par L’ARN polymérase II (Pol II)—dans tous les ciliés sauf; c’était l’exception unique des transcrits Pol II avant la publication de l’identification tr des plantes terrestres . En utilisant le motif télomère relativement long de L’Allium pour rechercher son TR dans le pool de données de séquence D’ARN total, Fajkus et al., (2019) ont montré qu’un ARN non codant précédemment caractérisé impliqué dans la réaction de stress chez A. thaliana, appelé AtR8, était bien la sous-unité d’ARN télomérase (Wu et al., 2012; Fajkus et coll., 2019). Il s’agissait d’une transcription de L’ARN polymérase III (Pol III) contenant les éléments régulateurs correspondants dans sa structure de promoteur. Pendant longtemps, les chercheurs s’attendaient à ce que plant TR soit si divergent qu’il serait impossible de l’identifier sur la base d’une recherche d’homologie (Cifuentes-Rojas et al., 2011)., Cependant, un certain degré de similitude a été utilisé avec succès pour identifier un TR commun chez plusieurs espèces D’Allium avec Blast comparatif. Étonnamment, l’homologie de séquence, la présence des mêmes éléments régulateurs et une région de gabarit correspondante ont conduit à l’identification de TRs chez Allium, Arabidopsis et plus de 70 autres plantes distantes, y compris celles avec des motifs télomères divergents comme Genlisea, Cestrum et Tulbaghia., Pour autant que nous sachions, il n’y a toujours pas de données sur une algue TR, ce qui permettrait d’élucider si la transcription Pol III de TR est une caractéristique générale pour toutes les plantes ou non. Ce travail (Fajkus et al., 2019), basé sur CRISPR knock-out et d’autres expériences, a également montré qu’un candidat d’ARN télomérase précédemment identifié chez A. thaliana (Cifuentes-Rojas et al., 2011; Beilstein et coll., 2012) n’était pas une sous-unité de modèle fonctionnelle de la télomérase, comme l’a également démontré peu après (Dew-Budd et al., 2019)., En supposant que la dépendance Pol II/Pol III pour la transcription de TR est un marqueur évolutif fiable, les recherches futures sur TR dans d’autres lignées principales d’eucaryotes ouvriront probablement de nouvelles perspectives sur l’origine des eucaryotes. Les gènes de la télomérase et les séquences des télomères sont des sources d’information non reconnues dans cette direction, et la découverte D’une voie de biogenèse tr dépendante de Pol III chez les lignées ciliées et végétales pourrait représenter les premières étapes dans cette direction (Greider et Blackburn, 1989; Fajkus et al., 2019).
comment les Chromosomes sont-ils devenus linéaires?,
Une grande majorité des procaryotes contiennent des chromosomes circulaires alors que les chromosomes linéaires sont la règle chez les eucaryotes., Par conséquent, il existe deux scénarios possibles dans lesquels (i) la linéarisation a été effectuée par une télomérase primitive, précédant d’autres processus qui ont conduit aux caractéristiques et fonctions chromosomiques linéaires actuelles ou (ii) la linéarisation d’un chromosome circulaire pré-eucaryote était initialement indépendante de la télomérase, mais juste avant la divergence des eucaryotes actuels, une télomérase primitive a commencé à occuper les extrémités des chromosomes et est devenue essentielle pour les chromosomes linéaires nouvellement formés (Nosek et al., 2006). Villasante et coll., (2007) ont proposé un scénario évolutif dans lequel la rupture du chromosome circulaire procaryote ancestral activait un mécanisme de transposition aux extrémités de l’ADN, permettant la formation de télomères par un mécanisme de réplication dépendant de la recombinaison: les conséquences de cette hypothèse ont conduit à la conclusion surprenante que les centromères eucaryotes étaient dérivés des télomères.
fait intéressant, le processus opposé à la linéarisation, c’est-à-dire la formation de chromosomes circulaires (également appelés chromosomes en anneau) a émergé de temps en temps au cours de l’évolution des eucaryotes, bien qu’étant très instable., Par exemple, dans le cas D’Amaranthus tuberculatus, les chromosomes cycliques sont apparus comme une réponse induite par le stress, portant une résistance à un herbicide (glyphosate); ces chromosomes cycliques supplémentaires n’ont pas montré d’hybridation avec des sondes télomères dans l’analyse du caryotype (Koo et al., 2018). Le système de télomérase presque universel et l’exceptionnalité des chromosomes circulaires chez les eucaryotes ne nous permettent pas de soutenir une hypothèse par rapport à l’autre., Cependant, la machinerie recombinationnelle utilisée dans l’allongement alternatif des télomères (ALT), une voie indépendante de la télomérase, associée à certains cancers humains (Zhang et al., 2019), est déjà présent chez les procaryotes. En outre, il existe des preuves de linéarisation chromosomique se produisant indépendamment dans des lignées distinctes de procaryotes (Ferdows et Barbour, 1989; Nosek et al., 1995; Volff et Altenbuchner, 2000). Par conséquent, l’hypothèse selon laquelle le premier chromosome eucaryote linéaire (provenant d’un ancêtre procaryote) était indépendant de la télomérase semble plus probable., Certains exemples montrent que le système à base de télomérase n’est pas essentiel pour le maintien des télomères chez tous les eucaryotes: rétrotransposons chez les télomères de Drosophile, répétitions satellites chez Chironomus, un autre insecte (Rubin, 1978; Biessmann et Mason, 2003), et ALT dans les cancers humains négatifs à la télomérase (Hu et al., 2016; Zhang et coll., 2019). Pourtant, certains de ces systèmes peuvent ne pas être aussi différents, et peuvent peut-être partager une origine commune: chez la drosophile, le maintien des télomères, basé sur la rétrotransposition, n’est pas trop distinct du mécanisme à base de télomérase (Danilevskaya et al.,, 1998), conduisant à l’hypothèse que la télomérase elle-même pourrait être un ancien rétrotransposon. Mais certainement, les espèces végétales à télomérase négative n’ont pas été découvertes à ce jour et toutes les exceptions, dans lesquelles le télomère typique de type végétal était absent, ont été montrées plus tard pour avoir des motifs différents, mais toujours synthétisés par télomérase. Néanmoins, la machinerie ALT est présente dans les plantes parallèlement à l’activité télomérase (Watson et Shippen, 2007; Ruckova et al., 2008). Des questions intéressantes sur le rôle de la télomérase, des télomères et leur maintien dans les tumeurs végétales en découlent., Un point intéressant concerne l’absence de métastases chez les plantes, malgré la présence d’ALT, peut-être liée à la rigidité des tissus végétaux ou à des systèmes immunitaires différents de ceux des animaux (Seyfried et Huysentruyt, 2013).
bien que nous acquérions de plus en plus de connaissances sur la biologie des télomères, nous sommes toujours incapables d’expliquer l’émergence de la télomérase chez les eucaryotes., Les preuves actuelles soutiennent l’hypothèse selon laquelle l’émergence des eucaryotes avec leurs chromosomes linéaires, leurs télomères et leur télomérase était liée à l’apparition d’introns splicéosomaux chez les hôtes archéens (Koonin, 2006; Fajkus et al., 2019). La similitude entre TERT et d’autres rétroéléments a été discutée pendant un certain temps (Pardue et al., 1997). Fait remarquable, une étude relativement récente a montré que TERT, en tant que membre probable des introns du groupe II de la descendance, est séquentiellement proche des rétrotransposons D’éléments de type Pénélope (Gladyshev et Arkhipova, 2007)., Mais TERT n’est que l’un des deux composants essentiels de la télomérase, et TR est, dans son origine, encore plus énigmatique en raison de sa faible conservation des séquences chez tous les eucaryotes .
Conclusion
Au début de l’ère de la génomique végétale, la séquence des télomères était considérée comme presque immuable. La conservation générale des télomères et du système de télomérase suggère que toutes les plantes peuvent avoir le télomère de type végétal tttaggg. L’identification de séquences de télomères inhabituelles dans des génomes végétaux complexes, dans de nombreux cas avec des valeurs c géantes (comme dans Cestrum et Allium sp.,), en valait la peine, car le motif exceptionnellement long Allium telomere était l’indice dans la recherche d’un véritable TR dans les plantes terrestres. La TR nouvellement décrite chez les plantes et d’autres recherches sur les télomères/télomérases dans les clades basaux d’algues pourraient révéler des informations précieuses sur l’évolution précoce, donc la recherche sur les télomères végétaux peut contribuer de manière significative à des hypothèses sur l’émergence des eucaryotes.
contributions des auteurs
VP et SG ont également contribué à la rédaction, à l’édition et à la préparation de cette mini-revue.,
Financement
conflit d’intérêts
Les auteurs déclarent que la recherche a été menée en l’absence de toute relation commerciale ou financière pouvant être interprétée comme un conflit d’intérêts potentiel.
Remerciements
d’Un Millier de Plantes Transcriptomes Initiative (2019). Un millier de transcriptomes végétaux et la phylogénomique des plantes vertes. La Nature 574 (7780), 679-685. doi: 10.1038/s41586-019-1693-2
PubMed Abstract | CrossRef Texte Intégral | Google Scholar
