Come si può vedere in Figura1, i nucleotidi variano solo leggermente, e solo nella base azotata. Nel caso del DNA, quelle basi sono adenina, guanina, citosina e timina. Si noti la somiglianza delle forme di adenina e guanina e anche la somiglianza tra citosina e timina. A e G sono classificati come purine, mentre C e T sono classificati come pirimidine. Finché nominiamo le cose, notate “desossiribosio” e”ribosio”. Come suggerisce il nome, il desossiribosio è solo un ribosio senza ossigeno., Più specificamente, dove c’è un gruppo idrossilico attaccato al 2-carbonio del ribosio, c’è solo un idrogeno attaccato al 2-carbonio del desossiribosio. Questa è l’unica differenza tra i due zuccheri.
Nella costruzione casuale di un singolo filamento di acido nucleico in vitro, non ci sono regole particolari riguardanti l’ordinamento dei nucleotidi rispetto alle loro basi. Le identità delle loro basi azotate sono irrilevanti perché i nucleotidi sono attaccati da legami fosfodiesterici attraverso il gruppo fosfato e il pentoso., È quindi spesso indicato come la spina dorsale di zucchero-fosfato. Se scomponiamo la parola “fosfodiestere”, vediamo che descrive abbastanza facilmente la connessione: gli zuccheri sono collegati da due legami estere ( —O—) con un fosforo in mezzo. Una delle idee che spesso confonde gli studenti è la direzionalità di questo legame, e quindi degli acidi nucleici in generale. Ad esempio, quando parliamo di DNA polimerasi, l’enzima che catalizza l’aggiunta di nucleotidi nelle cellule viventi, diciamo che funziona in una direzione da 5 primi (5’) a 3 primi (3’)., Questo può sembrare arcano molecolare-biologo-parlare, ma in realtà è molto semplice. Dai un’altra occhiata a due dei nucleotidi uniti dal legame fosfodiestere (Figura \(\PageIndex{1}\), in basso a sinistra). Un nucleotide dell’adenina è unito ad un nucleotide della citosina. Il legame fosfodiestere collegherà sempre il 5-carbonio di un desossiribosio (o ribosio nell’RNA) al 3-carbonio dello zucchero successivo. Ciò significa anche che su un’estremità di una catena di nucleotidi collegati, ci sarà un gruppo fosfato libero 5’ (- PO4) e sull’altra estremità, un idrossile libero 3’ (- OH)., Questi definiscono la direzionalità di un filamento di DNA o RNA.

Il DNA si trova normalmente come molecola a doppio filamento nella cellula mentre l’RNA è per lo più a filamento singolo., È importante capire però, che nelle condizioni appropriate, il DNA potrebbe essere fatto a singolo filamento, e RNA può essere a doppio filamento. In effetti, le molecole sono così simili che è persino possibile creare molecole ibride a doppio filamento con un filamento di DNA e uno di RNA. È interessante notare che le doppie eliche RNA-RNA e le doppie eliche RNA-DNA sono in realtà leggermente più stabili rispetto alla doppia elica DNA-DNA più convenzionale.,
La base della natura a doppio filamento del DNA, e in effetti la base degli acidi nucleici come mezzo per la conservazione e il trasferimento di informazioni genetiche, è l’accoppiamento di base. L’accoppiamento di base si riferisce alla formazione di legami idrogeno tra adenine e timine e tra guanina e citosina. Queste coppie sono significativamente più stabili di qualsiasi associazione formata con le altre possibili basi. Inoltre, quando queste associazioni base-coppia si formano nel contesto di due filamenti di acidi nucleici, la loro spaziatura è anche uniforme e altamente stabile., Si può ricordare che i legami idrogeno sono legami relativamente deboli. Tuttavia, nel contesto del DNA, il legame idrogeno è ciò che rende il DNA estremamente stabile e quindi adatto come mezzo di conservazione a lungo termine per l’informazione genetica. Poiché anche nei procarioti semplici, le doppie eliche del DNA sono lunghe almeno migliaia di nucleotidi, ciò significa che ci sono diverse migliaia di legami idrogeno che tengono insieme i due fili., Sebbene qualsiasi interazione di legame tra idrogeno da nucleotide a nucleotide possa facilmente essere temporaneamente interrotta da un leggero aumento della temperatura o da un minuscolo cambiamento nella forza ionica della soluzione, una doppia elica completa di DNA richiede temperature molto elevate (generalmente superiori a 90 ° C) per denaturare completamente la doppia elica in singoli filamenti.
Poiché esiste un accoppiamento esatto uno a uno di nucleotidi, risulta che i due filamenti sono essenzialmente copie di backup l’uno dell’altro-una rete di sicurezza nel caso in cui i nucleotidi vengano persi da un filamento., Infatti, anche se parti di entrambi i fili sono danneggiati, finché l’altro filo è intatto nell’area del danno, allora le informazioni essenziali sono ancora lì nella sequenza complementare del filo opposto e possono essere scritte in posizione. Tenete a mente però, che mentre un filamento di DNA può quindi agire come un” backup ” dell’altro, i due filamenti non sono identici – sono complementari. Una conseguenza interessante di questo sistema di fili complementari e antiparalleli è che i due fili possono portare ciascuna informazioni uniche.,
Le coppie di geni bidirezionali sono due geni su filamenti opposti di DNA, ma condividono un promotore, che si trova tra di loro. Poiché il DNA può essere fatto solo in una direzione, da 5 ‘ a 3’, questo promotore bidirezionale, spesso un’isola CpG (vedi capitolo successivo), invia quindi l’RNA polimerasi per ciascun gene in direzioni fisiche opposte. Questo è stato dimostrato per un certo numero di geni coinvolti nei tumori (seno, ovarico), ed è un meccanismo per coordinare l’espressione di reti di prodotti genici.
I filamenti di una doppia elica del DNA sono antiparalleli., Ciò significa che se osservassimo una doppia elica di DNA da sinistra a destra, un filamento sarebbe costruito nella direzione da 5’ a 3’, mentre il filamento complementare è costruito nella direzione da 3’ a 5’. Questo è importante per la funzione degli enzimi che creano e riparano il DNA, come discuteremo presto. Nella figura \ (\PageIndex{1}\), il filo sinistro è da 5 ‘a 3’ dall’alto verso il basso e l’altro è da 5’ a 3’ dal basso verso l’alto.
Dal punto di vista fisico, le molecole di DNA sono caricate negativamente (tutti quei fosfati) e normalmente una doppia elica con una torsione destrorsa., In questo stato normale (chiamato anche conformazione “B”), una torsione completa della molecola comprende 11 coppie di basi, con 0,34 nm tra ciascuna base nucleotidica. Ciascuna delle basi azotate sono planari, e quando accoppiato con la base complementare, forma un at planare “gradino” sulla “scala” del DNA. Questi sono perpendicolari all’asse longitudinale del DNA. La maggior parte del DNA fluttuante in una cellula, e la maggior parte del DNA in qualsiasi soluzione acquosa di osmolarità e pH quasi fisiologici, si trova in questa conformazione B., Tuttavia, sono state trovate altre conformazioni, di solito in circostanze ambientali molto specifiche. Una conformazione compressa, A-DNA, è stata osservata come un artefatto di cristallizzazione in vitro, con un po ‘ più basi per turno, una lunghezza di giro più corta e coppie di basi che non sono perpendicolari all’asse longitudinale. Un altro, Z-DNA, sembra formarsi transitoriamente in tratti di DNA ricchi di GC in cui, curiosamente, il DNA torce nella direzione opposta.
È stato suggerito che entrambe le forme A e Z del DNA sono, infatti, fisiologicamente rilevanti., Ci sono prove che suggeriscono che la forma A può verificarsi nelle doppie eliche ibride RNA-DNA e quando il DNA è complessato ad alcuni enzimi. La conformazione Z può verificarsi in risposta alla metilazione del DNA. Inoltre, la conformazione “normale” del B-DNA è qualcosa di una struttura idealizzata basata sull’essere completamente idratata, come è certamente molto probabile all’interno di una cellula. Tuttavia, questo stato di idratazione è in continua evoluzione, anche se minuziosamente, quindi la conformazione del DNA spesso varia leggermente dai parametri di conformazione B in Figura \(\PageIndex{2}\).,
Nei procarioti, il DNA si trova nel citoplasma (piuttosto ovvio poiché non c’è altra scelta in quei semplici organismi), mentre negli eucarioti, il DNA si trova all’interno del nucleo. Nonostante le differenze nelle loro posizioni, il livello di protezione dalle forze esterne e, soprattutto, le loro dimensioni, sia il DNA procariotico che quello eucariotico sono confezionati con proteine che aiutano a organizzare e stabilizzare la struttura cromosomica complessiva., Relativamente poco è compreso per quanto riguarda l’imballaggio cromosomico procariotico sebbene ci siano somiglianze strutturali tra alcune delle proteine trovate nei cromosomi procarioti ed eucariotici. Pertanto, la maggior parte dei corsi introduttivi di biologia cellulare si attengono al confezionamento cromosomico eucariotico.

Il DNA nudo, sia procariotico che eucariotico, è un filo di materiale estremamente sottile, di circa 11 nm di diametro. Tuttavia, date le dimensioni dei genomi eucariotici, se il DNA fosse immagazzinato in questo modo all’interno del nucleo, diventerebbe ingestibilmente aggrovigliato. Immagina un secchio in cui hai gettato un centinaio di metri di filo senza alcun tentativo di organizzarlo avvolgendolo o raggruppandolo., Ora considerare se si sarebbe in grado di raggiungere in quel secchio tirare su un filo, e si aspettano di tirare su un solo filo, o se invece si rischia di tirare su almeno un piccolo groviglio di filato. La cella fa essenzialmente quello che faresti con il filato per mantenerlo organizzato: è confezionato ordinatamente in matasse più piccole e gestibili. Nel caso del DNA, ogni cromosoma è avvolto attorno a un complesso istonico per formare il primo ordine di organizzazione cromosomica: il nucleosoma.
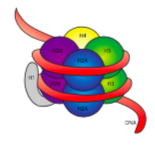
La fibra a 30 nm è tenuta insieme da due serie di interazioni. In primo luogo, l’istone linker, H1, riunisce i nucleosomi in una struttura approssimativa di 30 nm., Questa struttura viene quindi stabilizzata dai legami disolfuro che si formano tra l’istone H2A di un nucleosoma e l’istone H4 del suo vicino.
Gli istoni sono una famiglia di proteine di base (caricate positivamente). Tutti funzionano principalmente nell’organizzazione del DNA e il nucleosoma si forma quando il DNA avvolge (poco più di 2 volte) attorno a un nucleo di otto istoni – due ciascuno di H2A, H2B, H3 e H4. Il numero e la posizione delle cariche positive (principalmente da lisine e arginine) sono cruciali per la loro capacità di legare strettamente il DNA, che come precedentemente sottolineato, è molto caricato negativamente., Che” gli opposti si attraggono ” idea non è solo un incontri suggerimento dalle colonne di consulenza.

All’esame della struttura 3D del complesso del nucleo dell’istone, vediamo che mentre i domini di interazione proteica relativamente non caricati tengono gli istoni insieme al centro, i residui caricati positivamente si trovano intorno all’esterno del complesso, disponibili per interagire con i fosfati caricati negativamente del DNA.,
In un capitolo successivo, discuteremo di come gli enzimi leggono il DNA per trascrivere le sue informazioni su pezzi di RNA più piccoli e più gestibili. Per ora, abbiamo solo bisogno di essere consapevoli che in un dato momento, gran parte del DNA è confezionato ermeticamente, mentre alcune parti del DNA non lo sono. Poiché le parti che sono disponibili per l’uso possono variare a seconda di ciò che sta accadendo a/nella cellula in un dato momento, l’imballaggio del DNA deve essere dinamico. Ci deve essere un meccanismo per allentare rapidamente il legame del DNA agli istoni quando quel DNA è necessario per l’espressione genica e per stringere il legame quando non lo è., Come risulta, questo processo comporta acetilazione e deacetilazione degli istoni.

Le acetiltransferasi istoniche (HATs) sono enzimi che collocano un gruppo acetilico su una lisina di una proteina istonica. I gruppi acetilici sono caricati negativamente e l’acetilazione non solo aggiunge un gruppo caricato negativamente, ma rimuove anche la carica positiva dalla lisina. Ciò ha l’effetto non solo di neutralizzare un punto di attrazione tra la proteina e il DNA, ma anche di respingerlo leggermente (con cariche simili)., Dall’altro lato del meccanismo, le Istone Deactylases (HDACs) sono enzimi che rimuovono l’acetilazione e quindi ripristinano l’interazione tra la proteina dell’istone e il DNA. Poiché questi sono enzimi così importanti, è ovvio che non sono autorizzati a operare volenti o nolenti su qualsiasi istone disponibile, e infatti, si trovano spesso in un complesso con altre proteine che controllano e coordinano la loro attivazione con altri processi come l’attivazione della trascrizione.
