Introduzione
I telomeri sono strutture nucleoproteiche alle estremità dei cromosomi eucarioti lineari. Risolvono due grandi problemi finali allo stesso tempo. Il primo riguarda la protezione dell’estremità cromosomica. Si stima che le normali cellule umane debbano riparare almeno 50 interruzioni endogene a doppio filamento (DSB) per cellula per ciclo cellulare (Vilenchik e Knudson, 2003)., I telomeri distinguono le estremità cromosomiche naturali dai DSB dannosi e ne impediscono la riparazione ectopica, ad esempio mediante fusioni end-to-end di cromosomi (vanSteensel e deLange, 1997). Il secondo è il problema della replica finale che si occupa del mantenimento di lunghezze adeguate dei telomeri. Questo è stato riconosciuto indipendentemente da due ricercatori (Watson, 1972; Olovnikov, 1973). Poiché le DNA polimerasi DNA-dipendenti replicative non possono completare la sintesi del DNA alle estremità dei cromosomi, la compensazione per la perdita di sequenza telomerica replicativa deve provenire da una DNA polimerasi RNA-dipendente., Questo enzima, chiamato telomerasi, insieme alla prima sequenza di telomeri minisatelliti, è stato scoperto nella Tetraimena ciliata (Blackburn e Gall, 1978; Greider e Blackburn, 1985). Tuttavia, questo è solo un aspetto della manutenzione della lunghezza dei telomeri. Anche la regolazione epigenetica dell’omeostasi della lunghezza dei telomeri, compresa l’interazione di lunghe ripetizioni telomeriche non codificanti contenenti RNA e percorsi di attività esonucleasi, è stata ampiamente studiata a causa del suo potenziale terapeutico (Wellinger et al., 1996; Polotnianka et al., 1998; Pfeiffer e Lingner, 2012).,
La telomerasi, l’enzima incaricato di aggiungere sequenze di ripetizione dei telomeri all’estremità 3′ dei telomeri, è un enzima complesso conservato con numerosi componenti . In linea di principio, solo due componenti principali sono essenziali per l’attività enzimatica della telomerasi, un componente proteico cataliticamente attivo, chiamato transcriptasi inversa telomerica (TERT), e un componente modello, formato dalla subunità RNA telomerasi (TR). Mentre TERT è evolutivamente abbastanza ben conservato, TR è molto variabile, con lunghezze che vanno da ca. 150 nt (Tetrahymena) a più di 2.000 nt (funghi del genere Neurospora)., Solo una breve regione nell’intera molecola TR serve da modello per il DNA telomero appena sintetizzato (Greider e Blackburn, 1985; Qi et al., 2013). Questa regione in TR è solitamente formata da un motivo telomero completo seguito da uno parziale, quest’ultimo serve come regione di ricottura per il DNA telomero esistente. Sebbene, in linea di principio, sia necessario solo un singolo nucleotide extra (come motivo parziale), di solito ne viene trovato più di uno. Ad esempio, due nucleotidi extra formano il motivo di ricottura nei topi o cinque nell’uomo (Blasco et al., 1995; Feng et al., 1995)., Nelle piante, tuttavia, la dimensione della regione del modello è variabile, ad esempio, due in Arabidopsis thaliana, sette in Arabis sp. o sei in Nicotiana (Fajkus et al., 2019). Le altre regioni TR hanno funzioni interattive strutturali, normative e proteiche . Vedere anche una rappresentazione schematica della telomerasi e del suo ciclo di attività in Figura 1.

Figura 1 Rappresentazione schematica del ciclo di attività della telomerasi con il modello di telomero di tipo Arabidopsis. TERT, Telomero Trascrittasi inversa; TR, telomerasi RNA subunità., Figura basata su Sekhri (2014).
Quanto sono variabili le sequenze di telomeri?
Le sequenze di telomeri sono solitamente brevi minisatelliti disposti in modo tandem, tipicamente seguendo la formula (TxAyGz)n. La disposizione dei minisatelliti deriva dal modo in cui la telomerasi sintetizza il DNA, in breve, e motivi per lo più identici, uno per uno., Diverse ipotesi ritengono che tale disposizione sia importante perché promuove il riconoscimento di proteine specifiche dei telomeri da parte di omo ed eterodimeri e per il potenziale di formare quadruplessi G che possono stabilizzare le estremità dei cromosomi o servire come substrati per proteine specifiche dei telomeri (Spiegel et al., 2020; Tran et al., 2013). Le sequenze di telomeri sono ben conservate attraverso l’evoluzione, e grandi gruppi di organismi usano il motivo telomero tipico del gruppo per costruire il loro DNA telomero., Un numero gradualmente crescente di studi e proiezioni di grandi dimensioni hanno dimostrato che tutti i vertebrati testati e molti metazoani basali usano TTAGGG (Meyne et al., 1989; Traut et al., 2007) mentre Euarthropoda (artropodi), incluso Hexapoda (insetti), hanno TTAGG (Frydrychova et al., 2004; Vitkova et al., 2005). Costantemente, numerose eccezioni si stanno accumulando nel tempo, ad esempio, (A (G)1-8) in Dictyostelium (Emery e Weiner, 1981), TTAGGC in Ascaris lumbricoides (Nematoda) (Muller et al., 1991), TCAGG in coleotteri (coleotteri) (Mravinac et al., 2011), TAGGG/TAAGG/TAAGGG in Giardia (diplomonads) (Uzlikova et al.,, 2017), o TTNNNNAGGG in Yarrowia clade (lieviti) (Cervenak et al., 2019). Inoltre, i sistemi indipendenti dalla telomerasi, in cui la sequenza dei telomeri minisatelliti è stata persa e sostituita da ripetizioni complesse, sono rappresentati, ad esempio, da Ditteri e Chironomidi (recensiti in (Mason et al., 2016)). Per una revisione generale sulla sequenza di telomeri eucariotici vedi (Fajkus et al., 2005; Fulneckova et al., 2013).
La composizione dei telomeri nelle piante è ancora più diversificata., Qui usiamo il termine “piante” in senso lato, noto anche come Archaplastida o kingdom Plantae sensu lato, e comprende Rhodophyta (alghe rosse), Glaucophyta, il grado di alghe clorofite e il grado di alghe streptofite (complessivamente noto come alghe verdi) e Embryophyta (piante terrestri) (Iniziativa dei trascrittomi di mille piante, 2019). La sequenza tipica delle piante telomere è TTTAGGG, chiamata anche tipo Arabidopsis (o semplicemente tipo di pianta) poiché è stata scoperta in Arabidopsis thaliana (Richards e Ausubel, 1988) e ora in molte altre specie in quasi tutti gli ordini di piante., Sebbene TTTAGGG sia ancora il più frequente, vi è una significativa variabilità nelle sequenze di telomeri nei lignaggi algali rossi e verdi. Per quanto riguarda le alghe rosse (Rhodophyta), le informazioni sulla sequenza dei telomeri sono per lo più mancanti o frammentarie, anche se alcuni candidati telomeri sono stati scoperti in silico, come AATGGGGG per Cyanidioschyzon merolae (Nozaki et al., 2007), TTATT (T)AGGG per Galdieria sulphuraria (Fulneckova et al., 2013); TTAGGG è stato trovato in letture genomiche di Porphyra umbilicalis (Fulneckova et al., 2013), ma sono necessarie ulteriori prove per confermare la loro posizione terminale sui cromosomi., La diversità dei telomeri nelle alghe verdi riflette sia i cambiamenti dinamici che il suo carattere parafiletico. Anche se TTTAGGG prevale in Chlorophyta, come in generi Ostreococcus (Derelle et al., 2006) e Chlorella (Higashiyama et al., 1995), molti altri motivi divergenti sono stati rilevati anche lì, come TTAGGG nel genere Dunaliella e Stephanosphaeria (Fulneckova et al., 2012), e TTTTAGGG in Chlamydomonas (Petracek et al., 1990). In Streptophyta basale (Klebsormidiophyceae) sono stati descritti cambiamenti progressivi nei motivi da TTTAGGG a TTTTAGGG e TTTTAGG., La presenza di TTAGGG in Rhodophyta e Glaucophyta porta all’ipotesi che questo sia il motivo ancestrale nelle piante (Archaplastida) (Fulneckova et al., 2013).
Per quanto riguarda le piante terrestri, uno dei primi screening eseguiti ha mostrato che la sequenza di tipo Arabidopsis era la più comune e si conservava principalmente attraverso la loro filogenesi (Cox et al., 1993; Fuchs et al., 1995), anche se alcuni di questi autori avevano già rilevato diverse eccezioni nella famiglia Amaryllidaceae (ex Alliaceae), in cui la sequenza di tipo Arabidopsis era assente in diverse specie., Successivamente, la prima sequenza di telomeri insolita per le piante terrestri, il TTAGGG di tipo vertebrato, è stata caratterizzata nell’Aloe e in alcuni altri Asparagales (Weiss e Scherthan, 2002; Puizina et al., 2003; Sykorova et al., 2003c). È stata formulata un’ipotesi su perdite ripetute e recuperi della sequenza di telomeri TTTAGGG e TTAGGG in Asparagales (Adams et al., 2001). Con il postrefinimento dell’ordine Asparagales nell’APGIII (Angiosperm Phylogeny Group 2009) (Bremer et al.,, 2009), è stato dimostrato che si sono verificati solo due importanti interruttori evolutivi nella composizione della sequenza dei telomeri (piuttosto che diverse perdite e guadagni ripetuti), nel seguente ordine: il primo nella famiglia Iridaceae, in cui è avvenuto uno spostamento dal TTTAGGG di tipo vegetale al TTAGGG di tipo vertebrato, seguito dalle famiglie Xeronemataceae, Asphodelaceae e dal nucleo Asparagales (incluse Amarillidaceae s. l e Asparagaceae s.l.,); e la seconda all’interno della sottofamiglia Allioideae (precedentemente trattata come una famiglia separata, Alliaceae) in cui è emersa una sequenza di telomeri completamente nuova, CTCGGTTATGGG (Fajkus et al., 2016). Al di fuori delle Asparagales, nuove sequenze di telomeri sono state rilevate anche in gruppi di piante terrestri disparati come (i) Solanaceae, in cui è stata descritta la sequenza di telomeri di Cestrum elegans TTTTTTAGGG (Sykorova et al., 2003a; Sykorova et al., 2003b; Peska et al., 2008; Peska et al.,, 2015) e (ii) Lentibulariaceae, dove il genere Genlisea ha mostrato una notevole diversità con alcune specie caratterizzate dalle ripetizioni telomeriche di tipo Arabidopsis mentre altre mostravano varianti di sequenza mista TTCAGG e TTTCAGG (Tran et al., 2015).
Nonostante tutte le eccezioni dei motivi telomerici rilevate, la reale diversità delle sequenze telomeriche nelle piante terrestri è probabilmente molto sottovalutata. Una recente pubblicazione (Vitales et al.,, 2017), in cui è stata eseguita una selezione di sequenze di telomeri vegetali terrestri, ha scoperto che le sequenze di telomeri erano note chiaramente solo per meno del 10% delle specie e del 40% dei generi contenuti nel database delle piante rDNA (www.plantrdnadatabase.com), una risorsa che fornisce informazioni sulla citogenetica molecolare sulle piante terrestri (Garcia et al., 2012). Un riassunto della distribuzione della sequenza di telomeri nelle piante, dopo APG IV (The Angiosperm Phylogeny Group, 2016) (Byng et al., 2016), così come la più recente filogenesi vegetale (One Thousand Plant Transcriptomes Initiative, 2019) si trova in Figura 2.,
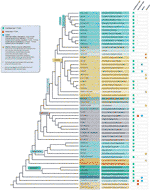
Figura 2 Motivi di telomeri in Archaplastida (piante in senso lato), basati sull’APG IV (The Angiosperm Phylogeny Group 2016) e sull’iniziativa One Thousand Plant Transcriptomes (2019). Le lunghezze dei rami non esprimono scale in tempo reale., Per semplicità e per risparmiare spazio, alcuni “gruppi” polifiletici (gradi) contrassegnati da un asterisco nell’albero sono stati rappresentati da un singolo ramo; per lo stesso motivo, diversi ordini minori (elencati nel quadrato blu nella parte superiore sinistra della figura) non sono raffigurati sull’albero. La prima etichetta di punta di solito si riferisce agli ordini di piante e in alcuni casi, a divisioni, gradi e persino famiglie; la seconda etichetta mostra famiglie rappresentative e in alcuni casi, ordini o generi rappresentativi.
Dalle proiezioni alla scoperta: come identificare i motivi telomerici?,
L’evidenza che una data sequenza candidata è una vera telomerica include diversi passaggi che dichiarano correttamente la sua localizzazione a tutti i termini cromosomici e, infine, il coinvolgimento della telomerasi nella sua sintesi. La citogenetica molecolare (principalmente mediante ibridazione in situ a fluorescenza, FISH) è diventata importante per visualizzare la localizzazione terminale di sonde etichettate di sequenze candidate a tutti i termini cromosomici. Tuttavia, PESCE standalone non è sufficiente per dimostrare la posizione molto terminale., Ad esempio, AcepSAT356 è stato proposto in cipolla come candidato telomero, sulla base dei risultati dell’analisi dei PESCI (Pich e Schubert, 1998). Tuttavia, la sua apparente posizione terminale in situ non è mai stata collegata in modo convincente alla funzione dei telomeri. In realtà, la scoperta della sequenza telomerica Allium minisatellite CTCGGTTATGGG e telomerasi significherebbe che AcepSAT356 è subterminale (Fajkus et al., 2019)., I segnali telomerici di PESCE positivi possono anche mascherare piccoli cambiamenti nei motivi dei telomeri come polimorfismi a singolo nucleotide o risultati falsi negativi possono derivare da telomeri brevi al di sotto del limite di rilevamento della tecnica.
Esistono due approcci aggiuntivi che determinano la posizione terminale a risoluzione maggiore rispetto a FISH; questi sono basati sull’attività dell’esonucleasi BAL31., La prima è la classica analisi del frammento di restrizione terminale (TRF), in cui i campioni trattati con BAL31 mostrano un progressivo accorciamento dei frammenti terminali e una diminuzione dell’intensità del segnale con l’aumentare del tempo di trattamento dell’esonucleasi. La successiva analisi delle lunghezze dei frammenti viene eseguita mediante ibridazione Southern-blot (Fojtova et al., 2015). Il secondo è genome skimming comparativo (dati NGS) del DNA genomico non digerito e digerito da BAL31, in parallelo., Nel set di dati trattati con BAL31, vi è una significativa sottorappresentazione delle sequenze telomeriche, pertanto le sequenze terminali sono identificate rispetto al set di dati non trattati, utilizzando strumenti bioinformatici RepeatExplorer o Tandem Repeats Finder .
L’altro test importante di una determinata sequenza di telomeri candidata in una specie è la dimostrazione dell’attività della telomerasi. In questo, un utile approccio sperimentale, sviluppato prima per le cellule umane, è il Telomero Repeat Amplification Protocol (TRAP) (Kim et al., 1994), seguito dal sequenziamento dei prodotti rilevati (Peska et al.,, 2015; Fajkus et al., 2016), che è un po ‘ meno sensibile ai risultati falsi positivi rispetto al PESCE. Tutti questi metodi, incluso il PESCE (Fuchs et al., 1995; Shibata e Hizume, 2011) e altri come l’ibridazione slot-blot (Sykorova et al., 2003c), e TRAPPOLA (Fulneckova et al., 2012; Fulneckova et al., 2016), può essere utilizzato per schermare i telomeri attraverso ampi gruppi di organismi complessi, comprese le piante., Tuttavia, solo una combinazione di metodi opportunamente scelti può portare in modo convincente a una conclusione sulla funzione telomerica di una sequenza candidata, poiché i risultati basati su un singolo approccio potrebbero essere fuorvianti. Una panoramica più completa delle strategie per l’identificazione della sequenza di candidati telomeri de novo, incluso il primo tentativo in Tetrahymena (Greider e Blackburn, 1985) sono riassunti in un articolo metodologico, con enfasi sull’approccio NGS utilizzato in piante con genomi estremamente grandi (Peska et al., 2017).
C’è omoplasia nelle sequenze di telomeri?,
Si pensa che la sequenza ancestrale dei telomeri sia TTAGGG ed è la più comune nell’albero della vita (Fulneckova et al., 2013). Tuttavia, sembra chiaro che la frequenza di omoplasia nell’evoluzione dei motivi dei telomeri è relativamente alta. Ad esempio, motivi brevi e semplici come il TTTAGGG di tipo vegetale sono apparsi indipendentemente e ripetutamente in cryptomonads, funghi oomiceti e alveolati; allo stesso modo, il TTAGGG di tipo vertebrato è emerso secondariamente in alcuni gruppi di piante (Asparagales, Rodophyta e alghe Chlorophyta) (Sykorova et al., 2003c; Fulneckova et al.,, 2012; Fulneckova et al., 2013; Somanathan e Baysdorfer, 2018). Il motivo per cui alcune sequenze di telomeri sono emerse più frequentemente di altre sequenze, di solito più complesse, è probabilmente correlato alle pressioni di selezione, che favorirebbero l’accuratezza per una particolare interazione DNA-proteina specifica della sequenza (Forstemann et al., 2003). Se ci fosse un cambiamento in ogni motivo telomerico, l’interferenza nella struttura nucleoproteica telomerica porterebbe necessariamente all’instabilità del genoma., Questa è la ragione per cui le sequenze di telomeri sono così evolutive stabili, comprendendo pochissime sequenze nuove e di successo, un modello coerente con l’idea di perdite ripetute e l’emergere delle tipiche sequenze di telomeri, come proposto per Asparagales (Adams et al., 2001).
La scoperta di omoplasia tra sequenze di telomeri solleva la domanda, quali sono le cause molecolari e i processi che avvengono durante questi spostamenti? Un cambiamento nella sequenza dei telomeri, nonostante sembri banale in alcuni casi (ad es.,, one extra T), può causare gravi interferenze con l’integrità del genoma, a causa di un equilibrio disturbato nelle interazioni DNA-proteine telomeri. Non è inoltre chiaro se un cambiamento nella sequenza dei telomeri possa avere qualche vantaggio evolutivo; a questo proposito, (Tran et al., 2015) ha suggerito che la comparsa di una citosina “metilabile” in un filamento di telomeri ricco di G aumenterebbe la possibilità di regolazione mediante modificazione epigenetica.
Quali sono le ragioni molecolari dei cambiamenti nei motivi dei telomeri?,
Per spiegare il cambiamento di sequenza dei telomeri, il primo candidato è la subunità modello della telomerasi, telomerasi RNA (TR). Il TR precedentemente identificato da lievito e vertebrati appartiene a un diverso gruppo di trascritti, la cui caratteristica di collegamento era che sono stati trascritti dalla RNA polimerasi II (Pol II)—in tutti tranne i ciliati; questa era la singola eccezione dai trascritti Pol II prima della pubblicazione dell’identificazione TR della pianta terrestre . Usando il motivo telomero relativamente lungo di Allium per cercare il suo TR all’interno del pool di dati della sequenza di RNA totale, Fajkus et al., (2019) ha dimostrato che un RNA non codificante precedentemente caratterizzato coinvolto nella reazione di stress in A. thaliana, chiamato AtR8, era effettivamente la subunità di RNA telomerasi (Wu et al., 2012; Fajkus et al., 2019). Era una trascrizione della RNA polimerasi III (Pol III) contenente i corrispondenti elementi regolatori nella sua struttura promotrice. Per molto tempo, i ricercatori si aspettavano che la pianta TR sarebbe stata così divergente che sarebbe stato impossibile identificarla sulla base di una ricerca omologica (Cifuentes-Rojas et al., 2011)., Tuttavia, un certo grado di somiglianza è stato utilizzato con successo per identificare un TR comune in diverse specie di Allium con esplosione comparativa. Sorprendentemente, l’omologia della sequenza, la presenza degli stessi elementi regolatori e una regione del modello corrispondente hanno portato all’identificazione di TRs in Allium, Arabidopsis e più di 70 altre piante lontanamente correlate, comprese quelle con motivi telomeri divergenti come Genlisea, Cestrum e Tulbaghia., Per quanto ne sappiamo, non ci sono ancora dati su qualsiasi TR algale, che chiarirebbe se la trascrizione di Pol III di TR è una caratteristica generale per tutte le piante o meno. Questo lavoro (Fajkus et al., 2019), basato su CRISPR knock-out e altri esperimenti, ha anche dimostrato che un candidato di RNA telomerasi precedentemente identificato in A. thaliana (Cifuentes-Rojas et al., 2011; Beilstein et al., 2012) non era una subunità funzionale del modello di telomerasi, come è stato anche dimostrato poco dopo da (Dew-Budd et al., 2019)., Supponendo che la dipendenza Pol II/Pol III per la trascrizione TR sia un marker evolutivo affidabile, la futura ricerca TR in altri principali lignaggi eucarioti aprirà probabilmente nuove intuizioni sull’origine degli eucarioti. I geni della telomerasi e le sequenze di telomeri sono fonti di informazione non riconosciute in questa direzione, e il ritrovamento di una via di biogenesi TR dipendente da Pol III in lignaggi ciliati e vegetali può rappresentare i primi passi in questa direzione (Greider e Blackburn, 1989; Fajkus et al., 2019).
In che modo i cromosomi sono diventati lineari?,
La stragrande maggioranza dei procarioti contiene cromosomi circolari mentre i cromosomi lineari sono la regola negli eucarioti., Quindi ci sono due possibili scenari in cui (i) la linearizzazione è stata eseguita da un primitivo telomerasi, che precede altri processi che hanno portato alla corrente lineare caratteristiche cromosomiche che funzioni, o (ii) la linearizzazione di un pre-eucariotica cromosoma circolare è stato inizialmente telomerasi indipendente, ma poco prima di corrente eucarioti discostato, un primitivo telomerasi iniziato a occupare le estremità dei cromosomi e divenne essenziale per la neonata cromosomi lineari (Nosek et al., 2006). Villasante et al., (2007) ha proposto uno scenario evolutivo in cui la rottura del cromosoma circolare procariotico ancestrale ha attivato un meccanismo di trasposizione alle estremità del DNA, consentendo la formazione di telomeri mediante un meccanismo di replicazione dipendente dalla ricombinazione: le conseguenze di questa ipotesi hanno portato alla sorprendente conclusione che i centromeri eucariotici sono derivati dai telomeri.
È interessante notare che il processo opposto alla linearizzazione, cioè la formazione di cromosomi circolari (chiamati anche cromosomi ad anello) è emerso di volta in volta durante l’evoluzione degli eucarioti, sebbene sia altamente instabile., Ad esempio, nel caso di Amaranthus tuberculatus, i cromosomi ad anello apparivano come una risposta indotta dallo stress, portando resistenza contro un erbicida (glifosato); questi cromosomi ad anello extra non mostravano ibridazione con sonde telomeriche nell’analisi del cariotipo (Koo et al., 2018). Il sistema di telomerasi quasi universale e l’eccezionalità dei cromosomi circolari negli eucarioti non ci permettono di sostenere un’ipotesi sull’altra., Tuttavia, il macchinario ricombinazionale utilizzato nell’allungamento alternativo dei telomeri (ALT), una via indipendente dalla telomerasi, associata a determinati tumori umani (Zhang et al., 2019), è già presente nei procarioti. Inoltre, vi è evidenza di linearizzazione cromosomica che si verifica indipendentemente in distinti lignaggi procarioti (Ferdows e Barbour, 1989; Nosek et al., 1995; Volff e Altenbuchner, 2000). Pertanto, l’ipotesi che il primo cromosoma eucariotico lineare (proveniente da un antenato procariote) fosse indipendente dalla telomerasi sembra più probabile., Ci sono alcuni esempi che mostrano che il sistema basato sulla telomerasi non è essenziale per il mantenimento dei telomeri in tutti gli eucarioti: retrotrasposoni nei telomeri di Drosophila, ripetizioni satellitari in Chironomus, un altro insetto (Rubin, 1978; Biessmann e Mason, 2003) e ALT nei tumori umani telomerasi-negativi (Hu et al., 2016; Zhang et al., 2019). Tuttavia, alcuni di questi sistemi potrebbero non essere così diversi e potrebbero forse condividere un’origine comune: in Drosophila, il mantenimento dei telomeri, basato sulla retrotrasposizione, non è troppo distinto dal meccanismo basato sulla telomerasi (Danilevskaya et al.,, 1998), portando all’ipotesi che la telomerasi stessa possa essere un precedente retrotrasposone. Ma certamente, le specie vegetali telomerasi-negative non sono state scoperte fino ad oggi e tutte le eccezioni, in cui il tipico telomero di tipo vegetale era assente, hanno successivamente dimostrato di avere motivi diversi, ma ancora sintetizzati dalla telomerasi. Tuttavia, il macchinario ALT è presente negli impianti in parallelo all’attività della telomerasi (Watson e Shippen, 2007; Ruckova et al., 2008). Da ciò derivano interessanti domande sul ruolo della telomerasi, dei telomeri e del loro mantenimento nei tumori vegetali., Un interessante riguarda l’assenza di metastasi nelle piante, nonostante la presenza di ALT, forse correlata alla rigidità del tessuto vegetale o a sistemi immunitari diversi rispetto agli animali (Seyfried e Huysentruyt, 2013).
Sebbene stiamo acquisendo una crescente conoscenza della biologia dei telomeri, non siamo ancora in grado di spiegare l’emergere della telomerasi negli eucarioti., Le prove attuali supportano l’ipotesi che l’emergere di eucarioti insieme ai loro cromosomi lineari, telomeri e telomerasi fosse correlato alla comparsa di introni spliceosomiali negli ospiti archaeali (Koonin, 2006; Fajkus et al., 2019). La somiglianza tra TERZ e altri retroelementi è stata discussa per qualche tempo (Pardue et al., 1997). Sorprendentemente, uno studio relativamente recente ha dimostrato che TERT, come probabile membro degli introni del progeny group II, è sequenzialmente vicino ai retrotrasposoni degli elementi simili a Penelope (Gladyshev e Arkhipova, 2007)., Ma TERT è solo uno dei due componenti essenziali della telomerasi e TR è, nella sua origine, ancora più enigmatico a causa della sua bassa conservazione della sequenza in tutti gli eucarioti .
Conclusione
All’inizio dell’era della genomica vegetale, la sequenza dei telomeri era considerata quasi immutabile. La conservazione generale dei telomeri e del sistema di telomerasi ha suggerito che tutte le piante possono avere il telomero di tipo vegetale TTTAGGG. L’identificazione di sequenze telomeriche insolite in genomi vegetali complessi, in molti casi con valori C giganti (come in Cestrum e Allium sp.,), valeva la pena, dal momento che il motivo telomero Allium eccezionalmente lungo era l’indizio nella ricerca di un vero TR nelle piante terrestri. La TR appena descritta nelle piante e ulteriori ricerche sui telomeri/telomerasi nei cladi basali delle alghe potrebbero rivelare preziose informazioni sull’evoluzione precoce, pertanto la ricerca sui telomeri vegetali può contribuire in modo significativo alle ipotesi sull’emergenza degli eucarioti.
Contributi dell’autore
VP e SG hanno contribuito allo stesso modo alla scrittura, all’editing e alla preparazione di questa mini-recensione.,
Finanziamento
Conflitto di interessi
Gli autori dichiarano che la ricerca è stata condotta in assenza di rapporti commerciali o finanziari che potrebbero essere interpretati come un potenziale conflitto di interessi.
Riconoscimenti
Iniziativa dei trascrittomi di mille piante (2019). Mille trascrittomi vegetali e la filogenomica delle piante verdi. Natura 574 (7780), 679-685. doi: 10.1038 / s41586-019-1693-2
PubMed Abstract / CrossRef Full Text / Google Scholar
