jak widać na Rys. 1, nukleotydy różnią się tylko nieznacznie i tylko w bazie azotowej. W przypadku DNA tymi zasadami są adenina, guanina, cytozyna i tymina. Zwróć uwagę na podobieństwo kształtów adeniny i guaniny, a także na podobieństwo cytozyny i tyminy. A i G są klasyfikowane jako puryny, podczas gdy C i T są klasyfikowane jako pirymidyny. Tak długo, jak nazywamy rzeczy, zwróć uwagę na „deoksyryboza” i „ryboza”. Jak sama nazwa wskazuje, deoksyryboza jest tylko rybozą bez tlenu., Dokładniej, gdy do 2-węgla rybozy przyłączona jest grupa hydroksylowa, do 2-węgla deoksyrybozy przyłączony jest tylko Wodór. To jedyna różnica między tymi dwoma cukrami.
w losowej budowie pojedynczej nici kwasu nukleinowego in vitro nie ma szczególnych zasad dotyczących kolejności nukleotydów w odniesieniu do ich zasad. Tożsamość ich zasad azotowych jest nieistotna, ponieważ nukleotydy są przyłączone przez wiązania fosfodiestrowe przez grupę fosforanową i pentozę., Jest więc często określany jako szkielet cukrowo-fosforanowy. Jeśli rozbijemy słowo „fosfodiester”, widzimy, że dość ręcznie opisuje połączenie: cukry są połączone dwoma wiązaniami estrowymi ( —O—) z fosforem pośrodku. Jednym z pomysłów, które często mylą uczniów, jest kierunkowość tego wiązania, a więc ogólnie kwasów nukleinowych. Na przykład, kiedy mówimy o polimerazie DNA, enzymie katalizującym dodawanie nukleotydów w żywych komórkach, mówimy, że działa ona w kierunku 5-prime (5′) do 3-prime (3′)., To może wydawać się tajemnicą biolog molekularny-mówić, ale to jest naprawdę bardzo proste. Spójrz jeszcze raz na dwa nukleotydy połączone wiązaniem fosfodiestrowym (rysunek \(\PageIndex{1}\), lewy dolny róg). Nukleotyd adeniny łączy się z nukleotydem cytozyny. Wiązanie fosfodiestrowe zawsze łączy 5-węgiel jednej deoksyrybozy (lub rybozy w RNA) z 3-węglem następnego cukru. Oznacza to również, że na jednym końcu łańcucha połączonych nukleotydów będzie wolna Grupa fosforanów 5′ (- PO4), a na drugim końcu wolna grupa hydroksylowa 3′ (- OH)., Definiują one kierunkowość nici DNA lub RNA.

DNA zwykle znajduje się jako dwuniciowa cząsteczka w komórce, podczas gdy RNA jest głównie jednoniciowa., Ważne jest jednak, aby zrozumieć, że w odpowiednich warunkach DNA może być jednoniciowe, a RNA może być dwuniciowe. W rzeczywistości cząsteczki są tak podobne, że możliwe jest nawet tworzenie dwuniciowych hybrydowych cząsteczek z jedną nicią DNA i jedną z RNA. Co ciekawe, podwójne helisy RNA – RNA i podwójne helisy RNA-DNA są w rzeczywistości nieco bardziej stabilne niż bardziej konwencjonalna podwójna helisa DNA-DNA.,
podstawą dwuniciowego charakteru DNA, a w rzeczywistości podstawą kwasów nukleinowych jako medium do przechowywania i przekazywania informacji genetycznej, jest parowanie zasad. Parowanie zasad odnosi się do tworzenia wiązań wodorowych między adeninami i tyminami oraz między guaninami i cytozynami. Pary te są znacznie bardziej stabilne niż jakikolwiek związek utworzony z innymi możliwymi bazami. Ponadto, gdy te związki zasad-par tworzą się w kontekście dwóch pasm kwasów nukleinowych, ich odstępy są również jednolite i wysoce stabilne., Można sobie przypomnieć, że wiązania wodorowe są relatywnie słabymi wiązaniami. Jednak w kontekście DNA wiązanie wodorowe jest tym, co sprawia, że DNA jest niezwykle stabilne i dlatego dobrze nadaje się jako długoterminowy nośnik informacji genetycznej. Ponieważ nawet u prostych prokariotów podwójne helisy DNA mają długość co najmniej tysięcy nukleotydów, oznacza to, że istnieje kilka tysięcy wiązań wodorowych trzymających oba pasma razem., Chociaż każda indywidualna interakcja wiązania wodorowego między nukleotydem a nukleotydem może zostać tymczasowo zakłócona przez niewielki wzrost temperatury lub niewielką zmianę siły jonowej roztworu, pełna podwójna helisa DNA wymaga bardzo wysokich temperatur (zazwyczaj powyżej 90oC), aby całkowicie denature podwójna helisa na poszczególne pasma.
ponieważ istnieje dokładne parowanie nukleotydów jeden do jednego, okazuje się, że dwie nici są zasadniczo kopiami zapasowymi siebie-siatką bezpieczeństwa w przypadku utraty nukleotydów z jednej nici., W rzeczywistości, nawet jeśli części obu nici są uszkodzone, tak długo, jak druga nić jest nienaruszona w obszarze uszkodzenia, to podstawowe informacje są nadal tam w komplementarnej sekwencji przeciwnej nici i mogą być zapisane na miejscu. Należy jednak pamiętać, że podczas gdy jedna nić DNA może w ten sposób działać jako „kopia zapasowa” drugiej, dwie nici nie są identyczne – są komplementarne. Interesującą konsekwencją tego systemu komplementarnych i antyporównawczych nici jest to, że każda z nich może przenosić unikalne informacje.,
dwukierunkowe pary genów to dwa geny na przeciwległych pasmach DNA, ale dzielące promotor, który leży pomiędzy nimi. Ponieważ DNA może być wykonane tylko w jednym kierunku, 5 'do 3′, Ten dwukierunkowy promotor, często Wyspa CpG (patrz następny rozdział), wysyła polimerazę RNA dla każdego genu w przeciwnych kierunkach fizycznych. Wykazano to dla wielu genów biorących udział w nowotworach (piersi, jajników) i jest mechanizmem koordynującym ekspresję sieci produktów genowych.
nici podwójnej helisy DNA są antyparallelowe., Oznacza to, że gdybyśmy spojrzeli na podwójną helisę DNA od lewej do prawej, jedna nić byłaby zbudowana w kierunku 5 'do 3′ , podczas gdy nić komplementarna jest zbudowana w kierunku 3′ do 5′. Jest to ważne dla funkcji enzymów, które tworzą i naprawiają DNA, o czym wkrótce będziemy dyskutować. Na rysunku \(\PageIndex{1}\) lewa nić wynosi 5′ do 3 'od góry do dołu, a druga 5 'do 3′ od dołu do góry.
z fizycznego punktu widzenia cząsteczki DNA są naładowane ujemnie (wszystkie te fosforany), a normalnie podwójna helisa z praworęcznym skręceniem., W tym normalnym stanie (zwanym również konformacją „B”), jeden pełny skręt cząsteczki obejmuje 11 par zasad, z 0,34 nm między każdą zasadą nukleotydu. Każda z zasad azotowych jest planarna, a po sparowaniu z zasadą uzupełniającą tworzy planarny „szczebel” na „drabinie” DNA. Są one prostopadłe do osi podłużnej DNA. Większość wolnego dna w komórce, a większość DNA w dowolnym roztworze wodnym o prawie fizjologicznej osmolarności i pH, Znajduje się w tej konformacji B., Jednak inne konformacje zostały znalezione, zwykle w bardzo specyficznych warunkach środowiskowych. Skompresowana konformacja, a-DNA, została zaobserwowana jako artefakt krystalizacji in vitro, z nieco większą liczbą zasad na obrót, krótszą długością obrotu i parami zasad, które nie są prostopadłe do osi podłużnej. Inny, z-DNA, wydaje się formować przejściowo w bogatych w GC odcinkach DNA, w których, co ciekawe, DNA skręca w przeciwnym kierunku.
sugerowano, że zarówno formy a, jak i z DNA są w rzeczywistości istotne fizjologicznie., Istnieją dowody sugerujące, że forma A może występować w hybrydowych podwójnych helisach RNA-DNA, a także gdy DNA jest kompleksowane do niektórych enzymów. Konformacja Z może wystąpić w odpowiedzi na metylację DNA. Co więcej, „normalna” konformacja B-DNA jest czymś w rodzaju wyidealizowanej struktury opartej na pełnym uwodnieniu, jak to jest z pewnością bardzo prawdopodobne wewnątrz komórki. Jednak ten stan nawodnienia ciągle się zmienia, choć drobiazgowo, więc konformacja DNA często będzie się nieznacznie różnić od parametrów konformacji B na rysunku \(\PageIndex{2}\).,
u prokariotów DNA znajduje się w cytoplazmie (raczej oczywiste, ponieważ nie ma innego wyboru w tych prostych organizmach), podczas gdy u eukariotów DNA znajduje się wewnątrz jądra. Pomimo różnic w ich lokalizacji, poziom ochrony przed siłami zewnętrznymi, a przede wszystkim ich wielkości, zarówno prokariotyczne, jak i eukariotyczne DNA jest pakowane z białkami, które pomagają organizować i stabilizować ogólną strukturę chromosomu., Stosunkowo niewiele rozumie się w odniesieniu do opakowania chromosomów prokariotycznych, chociaż istnieją podobieństwa strukturalne między niektórymi białkami znajdującymi się w chromosomach prokariotycznych i eukariotycznych. Dlatego większość kursów wprowadzających do biologii komórki trzyma się Opakowania chromosomów eukariotycznych.

nagie DNA, prokariotyczne lub eukariotyczne, jest bardzo cienką nicią materiału, około 11 nm średnicy. Jednak biorąc pod uwagę rozmiar genomów eukariotycznych, jeśli DNA byłoby przechowywane w ten sposób wewnątrz jądra, stałoby się niemoralnie splątane. Wyobraź sobie wiadro, do którego wrzuciłeś sto metrów przędzy bez jakiejkolwiek próby uporządkowania jej przez zwinięcie lub Wiązanie., Teraz zastanów się, czy będziesz w stanie sięgnąć do tego wiadra ciągnącego na jednym pasmie i oczekiwać, że podciągniesz tylko jedno pasmo, czy zamiast tego prawdopodobnie podciągniesz przynajmniej małą plątaninę przędzy. Komórka robi zasadniczo to, co zrobiłbyś z przędzy, aby utrzymać jej porządek: jest pakowana starannie w mniejsze, łatwe do zarządzania motki. W przypadku DNA, każdy chromosom jest zapętlony wokół kompleksu histonowego, tworząc pierwszy rząd organizacji chromosomalnej: nukleosom.
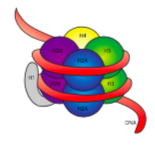
włókno 30-nm jest utrzymywane razem przez dwa zestawy interakcji. Po pierwsze, Histon łączący, H1, łączy nukleosomy w strukturę około 30 nm., Struktura ta jest następnie stabilizowana przez wiązania dwusiarczkowe, które tworzą się między histonem H2A jednego nukleosomu a histonem H4 jego sąsiada.
histony są rodziną podstawowych (dodatnio naładowanych) białek. Wszystkie one funkcjonują przede wszystkim w organizowaniu DNA, a nukleosom powstaje, gdy DNA owija się (nieco ponad 2 razy) wokół rdzenia ośmiu histonów-po dwa z H2A, H2B, H3 i H4. Liczba i położenie ładunków dodatnich (głównie z lizyn i arginin) są kluczowe dla ich zdolności do ścisłego wiązania DNA, które, jak wcześniej wspomniano, jest bardzo ujemnie naładowane., To, że” przeciwieństwa przyciągają ” pomysł nie jest tylko wskazówka randki z kolumn porad.

Po zbadaniu struktury 3D kompleksu rdzenia histonu, widzimy, że podczas gdy stosunkowo niezładowane domeny interakcji białkowych utrzymują histony razem w centrum, dodatnio naładowane pozostałości znajdują się na zewnątrz kompleksu, dostępne do interakcji z ujemnie naładowanymi fosforanami DNA.,
w późniejszym rozdziale omówimy, w jaki sposób enzymy odczytują DNA w celu transkrypcji jego informacji na mniejsze, łatwiejsze do opanowania fragmenty RNA. Na razie musimy tylko zdawać sobie sprawę, że w danym momencie znaczna część DNA jest szczelnie zapakowana, podczas gdy niektóre części DNA nie są. Ponieważ części, które są dostępne do użycia, mogą się różnić w zależności od tego, co dzieje się z / w komórce w danym momencie, opakowanie DNA musi być dynamiczne. Musi istnieć mechanizm szybkiego poluzowania wiązania DNA z histonami, gdy DNA jest potrzebne do ekspresji genów, i dokręcenia wiązania, gdy nie jest., Jak się okazuje, proces ten polega na acetylacji i deacetylacji histonów.

Acetylotransferazy Histonowe (HATs) są enzymami, które umieszczają grupę acetylową na lizynie białka histonowego. Grupy acetylowe są ujemnie naładowane, a acetylacja nie tylko dodaje ujemnie naładowaną grupę, ale także usuwa ładunek dodatni z lizyny. Ma to na celu nie tylko neutralizację punktu przyciągania między białkiem a DNA, ale nawet lekkie odpychanie go (z podobnymi ładunkami)., Po drugiej stronie mechanizmu Deaktylazy Histonowe (Hdac) są enzymami, które usuwają acetylację, a tym samym przywracają interakcję między białkiem histonowym a DNA. Ponieważ są to tak ważne enzymy, nie wolno im działać na żadnym dostępnym histonie, a w rzeczywistości często znajdują się w kompleksie z innymi białkami, które kontrolują i koordynują ich aktywację z innymi procesami, takimi jak aktywacja transkrypcji.
