wprowadzenie
telomery są strukturami nukleoproteinowymi na samych końcach liniowych chromosomów eukariotycznych. Rozwiązują dwa główne problemy końcowe w tym samym czasie. Pierwszy dotyczy ochrony końca chromosomu. Szacuje się, że normalne ludzkie komórki muszą naprawiać co najmniej 50 endogennych dwuniciowych przerw (DSBs) na komórkę na cykl komórkowy (Vilenchik and Knudson, 2003)., Telomery odróżniają naturalne końce chromosomów od szkodliwych DSBs i zapobiegają ich ektopowej naprawie, np. przez end-to-end fuzji chromosomów (vanSteensel and deLange, 1997). Drugi to problem replikacji końcowej, który zajmuje się utrzymaniem prawidłowych długości telomerów. Zostało to uznane niezależnie przez dwóch badaczy (Watson, 1972; Olovnikov, 1973). Ponieważ replikatywne polimerazy DNA zależne nie mogą zakończyć syntezy DNA na samych końcach chromosomów, kompensacja replikatywnej utraty sekwencji telomerów musi pochodzić z polimerazy DNA zależnej od RNA., Enzym ten, zwany telomerazą, wraz z pierwszą sekwencją minisatelitu telomerazy, został odkryty w tetrahymenie cyliatu (Blackburn and Gall, 1978; Greider and Blackburn, 1985). Jest to jednak tylko jeden z aspektów utrzymania długości telomerów. Epigenetyczna Regulacja homeostazy długości telomerów, w tym interakcja długich niekodujących powtórzeń telomerycznych zawierających RNA i szlaki aktywności egzonukleazy, były również szeroko badane ze względu na jej potencjał terapeutyczny (Wellinger et al., 1996; Polotnianka i in., 1998; Pfeiffer i Lingner, 2012).,
telomeraza, enzym odpowiedzialny za dodawanie sekwencji powtórzeń telomerów do końca 3′ telomerów, jest zachowanym kompleksowym enzymem z licznymi składnikami . Zasadniczo tylko dwa główne składniki są niezbędne do enzymatycznej aktywności telomerazy, katalizująco aktywny składnik białkowy, zwany telomerową odwrotną transkryptazą (TERT), oraz składnik wzorcowy, utworzony przez podjednostkę telomerazy RNA (TR). Podczas gdy TERT jest ewolucyjnie dość dobrze zachowany, TR jest bardzo zmienny, o długości od ok. 150 nt (Tetrahymena) do ponad 2000 nt (grzyby z rodzaju Neurospora)., Tylko krótki region w całej cząsteczce TR służy jako szablon dla nowo zsyntetyzowanego TELOMEROWEGO DNA (Greider and Blackburn, 1985; Qi et al., 2013). Ten region w TR jest zwykle utworzony przez kompletny motyw telomeru, a następnie częściowy, ten ostatni służy jako region wyżarzania dla istniejącego telomeru DNA. Chociaż w zasadzie potrzebny jest tylko jeden dodatkowy nukleotyd (jako motyw częściowy), zwykle znajduje się więcej niż jeden. Na przykład dwa dodatkowe nukleotydy tworzą motyw wyżarzania u myszy lub pięć u ludzi (Blasco et al., 1995; Feng et al., 1995)., U roślin wielkość regionu szablastego jest jednak zmienna, np. dwa U Arabidopsis thaliana, siedem u Arabis sp. lub sześć w Nicotiana (Fajkus et al., 2019). Pozostałe regiony TR mają funkcje strukturalne, regulacyjne i białkowe . Patrz również schematyczne przedstawienie telomerazy i jej cyklu aktywności na rysunku 1.

Rysunek 1 Schematyczne przedstawienie cyklu aktywności telomerazy za pomocą szablonu telomera typu Arabidopsis. TERT, telomeraza odwrotna Transkryptaza; TR, podjednostka telomerazy RNA., Rysunek na podstawie Sekhri (2014).
jak zmienne są sekwencje telomerów?
sekwencje telomerów są zwykle krótkimi minisatelitami ułożonymi tandemowo, Zwykle według wzoru (TxAyGz)N. układ minisatelitów pochodzi ze sposobu, w jaki telomeraza syntetyzuje DNA, w krótkich i najczęściej identycznych motywach, jeden po drugim., Kilka hipotez uważa, że taki układ jest ważny, ponieważ promuje rozpoznawanie telomerów specyficznych białek przez homo-i heterodimery oraz potencjał do tworzenia czworokątów G, które mogą stabilizować końce chromosomów lub służyć jako substraty dla telomerów specyficznych białek (Spiegel et al., 2020; Tran i in., 2013). Sekwencje telomerów są dobrze zachowane w procesie ewolucji, a duże grupy organizmów wykorzystują typowy dla grupy motyw telomerów do budowy telomerów DNA., Stopniowo rosnąca liczba badań i dużych badań przesiewowych wykazały, że wszystkie badane kręgowce i wiele podstawowych metazoanów używają TTAGGG (Meyne et al., 1989; Traut et al., 2007), podczas gdy Euarthropoda (stawonogi), w tym Heksapoda (owady), mają TTAGG (Frydrychova et al., 2004; Vitkova et al., 2005). Stopniowo, liczne wyjątki kumulują się w czasie ,np. (A (G) 1-8) w Dictyostelium (Emery and Weiner, 1981), TTAGGC w Ascaris lumbricoides (Nematoda) (Muller et al., 1991), TCAGG in Coleoptera (beetles) (Mravinac et al., 2011), TAGGG/TAAGG/TAAGG in Giardia (Diplomatonads) (Uzlikova et al.,, 2017), lub TTNNNAGGG in Yarrowia clade (yeasts) (Cervenak et al., 2019). Ponadto systemy niezależne od telomerazy, w których Sekwencja telomerów minisatelitu została utracona i zastąpiona przez złożone powtórzenia, są reprezentowane na przykład przez Diptera i Chironomidae (reviewed in (Mason et al., 2016)). Aby uzyskać ogólny przegląd sekwencji telomerów eukariotycznych patrz (Fajkus et al., 2005; Fulneckova et al., 2013).
skład telomerów u roślin jest jeszcze bardziej zróżnicowany., Tutaj używamy terminu” rośliny ” w szerokim znaczeniu, znanego również jako Archaeplastida lub kingdom Plantae sensu lato, i obejmującego Rhodophyta( czerwone algi), Glaucophyta, gatunek glonów Chlorofitowych i Gatunek glonów Streptofitowych (ogólnie znany jako zielone algi) i Embriophyta (rośliny lądowe) (inicjatywa tysiąca Transkryptomów roślin, 2019). Typową sekwencją roślin telomerowych jest TTTAGG, zwany także Arabidopsis-type (lub po prostu-plant-type), ponieważ został odkryty w Arabidopsis thaliana (Richards and Ausubel, 1988), a obecnie w wielu innych gatunkach w prawie wszystkich rzędach roślin., Chociaż TTTAGG jest nadal najczęstszy, istnieje znaczna zmienność sekwencji telomerów w czerwonych i zielonych liniach glonów. Jeśli chodzi o czerwone algi (Rhodophyta), informacje o sekwencji telomerów są w większości brakujące lub fragmentaryczne, chociaż niektóre kandydatury telomerów zostały odkryte w silico, takie jak AATGGGGG dla Cyanidioschyzon merolae (Nozaki et al., 2007), TTATT(T) AGGG dla Galdieria sulphuraria (Fulneckova et al., 2013); TTAGG został znaleziony w genomicznych odczytach Porphyra umbilicalis (Fulneckova et al., 2013), ale potrzeba więcej dowodów, aby potwierdzić ich końcowe położenie na chromosomach., Różnorodność telomerów w zielonych glonach odzwierciedla zarówno dynamiczne zmiany, jak i jej parafiletyczny charakter. Chociaż TTTAGG przeważa w Chlorophyta, takich jak w rodzaju Ostreococcus (Derelle et al., 2006) i Chlorella (Higashiyama et al., 1995), wykryto tam również wiele innych rozbieżnych motywów, takich jak TTAGG w rodzaju Dunaliella i Stephanosphaeria (Fulneckova et al., 2012) i TTTAGGG w Chlamydomonas (Petracek et al., 1990). U Streptophyta podstawnego (Klebsormidiophyceae)opisano postępujące zmiany motywów z TTTAGG do TTTAGG i TTTAGG., Obecność TTAGGG u Rhodophyta i Glaucophyta prowadzi do hipotezy, że jest to motyw przodków u roślin (Archaeplastida) (Fulneckova et al., 2013).
w odniesieniu do roślin lądowych, jedna z pierwszych przeprowadzonych ekranizacji wykazała, że sekwencja rodzaju Arabidopsis była najczęstsza i została zachowana głównie przez ich filogenezę (Cox et al., 1993; Fuchs et al., 1995), chociaż niektórzy z tych autorów wykryli już kilka wyjątków w rodzinie Amaryllidaceae (dawniej Alliaceae), w której Sekwencja rodzaju Arabidopsis była nieobecna u kilku gatunków., Później pierwszą nietypową dla roślin lądowych sekwencję telomerów, Typ kręgowców TTAGG, scharakteryzowano u aloesu i niektórych innych Szparagowców (Weiss i Scherthan, 2002; Puizina et al., 2003; Sykorova i in., 2003c). Sformułowano hipotezę o powtarzających się stratach i odzyskiwaniu sekwencji telomerów TTTAGG i ttaggg w Asparagales (Adams et al., 2001). Pozycja w obrębie rzędu Szparagowców (Asparagales) w obrębie APGIII (Angiosperm Phylogeny Group 2009) (Bremer et al.,, 2009), wykazano, że tylko dwa główne ewolucyjne przełączniki w składzie sekwencji telomerów wystąpiły (zamiast kilku powtarzających się strat i zysków), w następującej kolejności: pierwszy w rodzinie Iridaceae, w której nastąpiła zmiana z roślinnego typu TTTAGG do kręgowego typu TTAGG, a następnie rodziny Xeronemataceae, Asphodelaceae i rdzeniowce Asparagales (w tym Amarillidaceae s.L i Asparagaceae s. L.,), a drugi w obrębie podrodziny Allioideae (dawniej traktowanej jako osobna rodzina, Alliaceae), w której pojawiła się zupełnie nowa sekwencja telomerów, Ctcggttatgg (Fajkus et al., 2016). Poza Asparagales nowe sekwencje telomerów zostały również wykryte w lądowych grupach roślin jako odrębne jak (i) Solanaceae, w których opisano sekwencję telomerów Cestrum elegans TTTTAGG (Sykorova et al., 2003a; Sykorova i in., 2003b; Peska i in., 2008; Peska i in.,, 2015) I (ii) Lentibulariaceae, gdzie rodzaj Genlisea wykazywał niezwykłą różnorodność z niektórymi gatunkami charakteryzującymi się powtórzeniami telomerów z rodzaju Arabidopsis, podczas gdy inne wykazywały mieszane warianty sekwencji TTCAGG i Tttcagg (Tran et al., 2015).
pomimo wszystkich wykrytych wyjątków od motywu telomerów, prawdziwa różnorodność sekwencji telomerów u roślin lądowych jest prawdopodobnie znacznie niedoceniana. Ostatnia publikacja (Vitales et al.,, 2017), w którym przeprowadzono przesiewanie sekwencji telomerów roślin lądowych, stwierdzono, że sekwencje telomerów były znane tylko dla mniej niż 10% gatunków i 40% rodzajów zawartych w bazie danych roślin rDNA (www.plantrdnadatabase.com), zasób dostarczający informacji o cytogenetyce molekularnej roślin lądowych (Garcia et al., 2012). A summary of telomere sequence distribution in plants, following APG IV (The Angiosperm Phylogeny Group, 2016) (Byng et al., 2016), a także najnowszą filogenezę roślin (One Thousand Plant Transcriptomes Initiative, 2019) znajduje się na rysunku 2.,
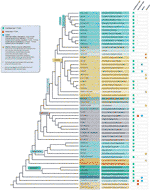
Rysunek 2 Motywy telomerów w Archaeplastida (rośliny w szerokim znaczeniu), oparte na systemie APG IV (The Angiosperm Phylogeny Group 2016) oraz na inicjatywie One Thousand Plant Transcriptomes (2019). Długości gałęzi nie wyrażają skal czasu rzeczywistego., Dla uproszczenia i zaoszczędzenia miejsca, niektóre polifiletyczne „grupy” (stopnie) oznaczone gwiazdką na drzewie zostały reprezentowane przez jedną gałąź; z tego samego powodu, kilka mniejszych rzędów (wymienionych w niebieskim kwadracie po lewej górnej stronie rysunku) nie są przedstawione na drzewie. Pierwsza Etykieta końcówki zwykle odnosi się do rzędów roślin, a w kilku przypadkach do podziałów, stopni, a nawet rodzin; druga Etykieta wyświetla rodziny reprezentatywne, a w kilku przypadkach, rzędy przedstawicielskie lub rodzaje.
od ekranizacji do odkrycia: jak można zidentyfikować motywy Telomeryczne?,
dowód na to, że dana Sekwencja kandydująca jest prawdziwą telomeryczną zawiera kilka kroków, które prawidłowo deklarują jej lokalizację na wszystkich końcach chromosomów, a ostatecznie udział telomerazy w jej syntezie. Cytogenetyka molekularna (głównie przez fluorescencję in situ hybrydyzacji, FISH) stała się ważna dla wizualizacji terminalnej lokalizacji znakowanych sond sekwencji kandydujących na wszystkich chromosomalnych końcach. Jednak samodzielne ryby to nie wystarczy, aby udowodnić bardzo terminal pozycji., Na przykład AcepSAT356 został zaproponowany w cebuli jako kandydat telomerów, na podstawie wyników analizy ryb (Pich i Schubert, 1998). Niemniej jednak jego pozorna lokalizacja terminala przez In situ nigdy nie została przekonująco powiązana z funkcją telomera. W rzeczywistości odkrycie Allium minisatelitu sekwencji telomerów CTCGGTTATGGG i telomerazy oznaczałoby, że AcepSAT356 jest subterminalny (Fajkus et al., 2019)., Pozytywne sygnały TELOMERYCZNE ryb mogą również maskować drobne zmiany w motywach telomerów, takich jak polimorfizmy pojedynczego nukleotydu lub fałszywie ujemne wyniki mogą wynikać z krótkich telomerów znajdujących się poniżej granicy wykrywalności techniki.
istnieją dwa dodatkowe podejścia, które określają pozycję końcową przy większej rozdzielczości niż ryby; są one oparte na aktywności egzonukleazy BAL31., Pierwszą z nich jest analiza klasycznego fragmentu ograniczenia terminali (TRF), w której próbki poddane działaniu BAL31 wykazują postępujące skracanie fragmentów terminali i spadek natężenia sygnału wraz ze wzrostem czasu leczenia egzonukleazą. Późniejsza analiza długości fragmentów jest wykonywana przez hybrydyzację Southern-blot (Fojtova et al., 2015). Drugi jest porównawczy Genom skimming (dane NGS) nondigested i BAL31-trawione genomic DNA, równolegle., W zestawie danych traktowanych przez BAL31 występuje znaczna niedostateczna reprezentacja sekwencji telomerów, dlatego sekwencje końcowe są identyfikowane przez porównanie z nieleczonym zestawem danych, przy użyciu narzędzi bioinformatycznych RepeatExplorer lub Tandem Repeats Finder .
innym ważnym testem danej sekwencji telomerów u danego gatunku jest wykazanie aktywności telomerazy. W tym, użytecznym podejściem eksperymentalnym, opracowany jako pierwszy dla komórek ludzkich, jest Telomere Repeat Amplification Protocol (TRAP) (Kim et al., 1994), a następnie sekwencjonowanie wykrytych produktów (Peska et al.,, 2015; Fajkus i in., 2016), która jest nieco mniej wrażliwa na wyniki fałszywie dodatnie niż ryba. Wszystkie te metody, w tym ryby (Fuchs et al., 1995; Shibata and Hizume, 2011) i inne, takie jak hybrydyzacja slot-blot (Sykorova et al., 2003c) oraz TRAP (Fulneckova et al., 2012; Fulneckova et al., 2016), może być stosowany do przesiewania telomerów w szerokich grupach złożonych organizmów, w tym roślin., Jednak tylko kombinacja odpowiednio dobranych metod może w przekonujący sposób doprowadzić do wniosku o funkcji telomeru sekwencji kandydackiej, ponieważ wyniki oparte na jednym podejściu mogą być mylące. Pełniejszy przegląd strategii de novo telomere candidate Sequence identification, w tym pierwszej próby Tetrahymena (Greider and Blackburn, 1985) podsumowano w artykule metodologicznym, z naciskiem na podejście NGS stosowane w roślinach o bardzo dużych genomach (Peska et al., 2017).
czy w sekwencjach telomerów jest Homoplazja?,
przodkowa Sekwencja telomerów jest uważana za TTAGGG i jest najczęściej spotykana w drzewie życia (Fulneckova et al., 2013). Wydaje się jednak jasne, że częstotliwość homoplazji w ewolucji motywu telomerów jest stosunkowo wysoka. Na przykład krótkie, proste motywy, takie jak tttagg roślin pojawiły się niezależnie i wielokrotnie w cryptomonads, oomycete grzybów i pęcherzyków; podobnie, kręgowców typu ttagg pojawił się wtórnie w niektórych grupach roślin (Asparagales, Rodophyta i glony Chlorophyta) (Sykorova et al., 2003C; Fulneckova i in.,, 2012; Fulneckova et al., 2013; Somanathan and Baysdorfer, 2018). Powód, dla którego niektóre sekwencje telomerów pojawiły się częściej niż inne, zwykle bardziej złożone sekwencje, jest prawdopodobnie związany z presją selekcji, co sprzyjałoby dokładności dla konkretnej sekwencji specyficznej interakcji DNA-białko (Forstemann et al., 2003). Jeśli nastąpiła zmiana w każdym motywie telomerów, ingerencja w strukturę nukleoprotein telomerycznych musiałaby prowadzić do niestabilności genomu., Jest to powód, dla którego sekwencje telomerów są tak stabilne ewolucyjnie, obejmujące bardzo mało nowatorskich i udanych sekwencji, wzór zgodny z ideą powtarzających się strat i pojawieniem się typowych sekwencji telomerów, jak zaproponowano dla Asparagales (Adams et al., 2001).
odkrycie homoplazji w sekwencjach telomerów rodzi pytanie, Jakie są molekularne przyczyny i procesy zachodzące podczas tych zmian? Zmiana sekwencji telomerów, mimo że w niektórych przypadkach wydaje się trywialna (np.,, jeden dodatkowy T), może powodować poważne zakłócenia w integralności genomu, z powodu zaburzenia równowagi w interakcjach telomerów DNA-białko. Nie jest również jasne, czy zmiana w sekwencji telomerów może mieć jakąkolwiek ewolucyjną przewagę; w tym względzie (Tran et al., 2015) zasugerował, że pojawienie się” metylowalnej ” cytozyny w bogatej w G nici telomeru podniesie możliwość regulacji przez modyfikację epigenetyczną.
jakie są molekularne przyczyny zmian motywów telomerów?,
aby wyjaśnić zmianę sekwencji telomerów, pierwszym kandydatem jest podjednostka wzorcowa telomerazy, telomeraza RNA (TR). Wcześniej zidentyfikowane TR od drożdży i kręgowców należy do innej grupy transkryptów, których cechą łączącą było to, że zostały one transkrybowane przez polimerazę RNA II (Pol II)—we wszystkich poza cyliatami; był to pojedynczy wyjątek od transkryptów Pol II przed publikacją identyfikacji TR roślin lądowych . Wykorzystując stosunkowo długi motyw telomeru Allium do poszukiwania jego TR w całkowitej puli danych sekwencji RNA, Fajkus et al., (2019) wykazał, że wcześniej scharakteryzowany noncoding RNA zaangażowany w reakcję stresową u A. thaliana, zwany AtR8, był rzeczywiście podjednostką telomerazy RNA (Wu et al., 2012; Fajkus i in., 2019). Była to transkrypcja polimerazy RNA III (Pol III) zawierającej odpowiednie elementy regulacyjne w swojej strukturze promotora. Przez długi czas naukowcy oczekiwali, że TR roślin będzie tak rozbieżne, że niemożliwe będzie zidentyfikowanie go na podstawie wyszukiwania homologicznego (Cifuentes-Rojas et al., 2011)., Jednak pewien stopień podobieństwa został z powodzeniem wykorzystany do identyfikacji TR pospolitego u kilku gatunków Allium o porównawczym Blastu. Co zaskakujące, homologia sekwencji, obecność tych samych elementów regulacyjnych i odpowiadający im region szablonu doprowadziły do identyfikacji TRs u Allium, Arabidopsis i ponad 70 innych odległych roślin, w tym tych z różnymi motywami telomerów, takimi jak Genlisea, Cestrum i Tulbaghia., O ile nam wiadomo, nadal nie ma danych na temat jakichkolwiek glonów TR, które wyjaśniałyby, czy transkrypcja Pol III TR jest cechą ogólną dla wszystkich roślin, czy też nie. Ta praca (Fajkus et al., 2019), w oparciu o CRISPR knock-out i inne eksperymenty, wykazały również, że wcześniej zidentyfikowany kandydat na telomerazę RNA u A. thaliana (Cifuentes-Rojas et al., 2011; Beilstein i in., 2012) nie był funkcjonalną podjednostką szablonu telomerazy, co również wykazano wkrótce po (Dew-Budd et al., 2019)., Zakładając, że zależność Pol II/Pol III dla transkrypcji TR jest wiarygodnym markerem ewolucyjnym, przyszłe badania TR w innych głównych liniach eukariotycznych prawdopodobnie otworzą nowe spojrzenie na pochodzenie eukariotów. Geny telomerazy i sekwencje telomerów są nierozpoznanymi źródłami informacji w tym kierunku, a odkrycie zależnego od POL III szlaku biogenezy TR w liniach ciliate i roślinnych może reprezentować pierwsze kroki w tym kierunku (Greider and Blackburn, 1989; Fajkus et al., 2019).
jak chromosomy stały się liniowe?,
zdecydowana większość prokariotów zawiera chromosomy koliste, podczas gdy chromosomy liniowe są regułą u eukariotów., Dlatego istnieją dwa możliwe scenariusze, w których albo (i) linearyzacja została przeprowadzona przez prymitywną telomerazę, poprzedzając inne procesy, które doprowadziły do obecnych liniowych chromosomów cechy i funkcje lub (ii) linearyzacja Pre-eukariotycznych kolistych chromosomów była początkowo telomerazy niezależne, ale tuż przed obecnymi eukariotyczne rozbieżne, prymitywna telomeraza zaczęła zajmować końce chromosomów i stała się niezbędna dla nowo powstałych liniowych chromosomów (Nosek et al., 2006). Villasante et al., (2007) zaproponował scenariusz ewolucyjny, w którym pęknięcie prokariotycznego kolistego chromosomu aktywowało mechanizm transpozycji na końcach DNA, umożliwiając tworzenie telomerów przez mechanizm replikacji zależnej od rekombinacji: konsekwencje tej hipotezy doprowadziły do zaskakującego wniosku, że eukariotyczne centromery pochodzą z telomerów.
Co ciekawe, proces odwrotny do linearyzacji, tj. tworzenie chromosomów kolistych (zwanych również chromosomami pierścieniowymi) pojawił się od czasu do czasu podczas ewolucji eukariotów, chociaż jest wysoce niestabilny., Na przykład, w przypadku Amaranthus tuberculatus, chromosomy pierścieniowe pojawiły się jako reakcja wywołana stresem, niosąc odporność na herbicyd (glifosat); te dodatkowe chromosomy pierścieniowe nie wykazały hybrydyzacji z sondami telomerowymi w analizie kariotypu (Koo et al., 2018). Niemal uniwersalny system telomerazy i wyjątkowość kolistych chromosomów u eukariotów nie pozwalają na poparcie jednej hipotezy nad drugą., Jednak rekombinacyjne Maszyny stosowane w alternatywnym wydłużeniu telomerów (ALT), telomerazy niezależny szlak, związane z niektórych ludzkich nowotworów (Zhang et al., 2019), występuje już u prokariotów. Ponadto istnieją dowody linearyzacji chromosomów występujących niezależnie w różnych liniach prokariotycznych (Ferdows and Barbour, 1989; Nosek et al., 1995; Volff i Altenbuchner, 2000). Dlatego bardziej prawdopodobna wydaje się hipoteza, że pierwszy liniowy chromosom eukariotyczny (pochodzący od przodka prokariotycznego) był niezależny od telomerazy., Istnieje kilka przykładów, które pokazują, że system oparty na telomerach nie jest niezbędny do utrzymania telomerów u wszystkich eukariotów: retrotransposony u telomerów Drosophila, powtórzenia satelitów u Chironomus, innego owada (Rubin, 1978; Biessmann i Mason, 2003)i ALT w nowotworach ludzkich bez telomerazy (Hu et al., 2016; Zhang et al., 2019). Jednak niektóre z tych systemów mogą nie być tak różne i być może mają wspólne pochodzenie: w Drosophila, utrzymanie telomerów, oparte na retrotransmisji, nie jest zbyt różni się od mechanizmu opartego na telomerazie (Danilevskaya et al.,, 1998), co prowadzi do hipotezy, że sama telomeraza może być dawnym retrotranspozonem. Ale z pewnością, gatunki roślin telomerazy ujemne nie zostały odkryte do tej pory i wszystkie wyjątki, w których typowa telomerazy typu roślinnego był nieobecny, wykazano później, że mają różne, ale nadal telomerazy syntetyzowane motywy. Niemniej jednak, mechanizm ALT jest obecny w roślinach równolegle do aktywności telomerazy (Watson and Shippen, 2007; Ruckova et al., 2008). Z tego wynikają interesujące pytania dotyczące roli telomerazy, telomerów i ich utrzymania w nowotworach roślinnych., Atrakcyjny jest brak przerzutów u roślin, pomimo obecności alat, być może związanej z sztywnością tkanek roślinnych lub innym układem odpornościowym niż u zwierząt (Seyfried i Huysentruyt, 2013).
chociaż zdobywamy coraz większą wiedzę na temat biologii telomerów, wciąż nie jesteśmy w stanie wyjaśnić pojawienia się telomerazy u eukariotów., Obecne dowody potwierdzają hipotezę, że pojawienie się eukariotów wraz z ich liniowymi chromosomami, telomerami i telomerazą było związane z pojawieniem się spliceosomalnych intronów w archaealnych gospodarzach (Koonin, 2006; Fajkus et al., 2019). Podobieństwo między TERT i innymi retroelementami jest omawiane od pewnego czasu (Pardue et al., 1997). Co ciekawe, stosunkowo niedawne badania wykazały, że TERT, jako prawdopodobny członek intronów grupy potomnej II, jest sekwencyjnie zbliżony do retrotranspozonów pierwiastka Penelopowego (Gladyshev and Arkhipova, 2007)., Ale TERT jest tylko jednym z dwóch podstawowych składników telomerazy, A TR jest, w swoim pochodzeniu, jeszcze bardziej enigmatyczny ze względu na jego niską sekwencję zachowania we wszystkich eukariotach .
podsumowanie
na początku ery genomiki roślin Sekwencja telomerów była uważana za niemal niezmienną. Ogólna konserwacja telomerów i systemu telomerazy sugerowała, że wszystkie rośliny mogą mieć telomery typu tttaggg. Identyfikacja nietypowych sekwencji telomerów w złożonych genomach roślinnych, w wielu przypadkach z wielkimi wartościami C (np. w Cestrum i Allium sp.,), było warte wysiłku, ponieważ wyjątkowo długi motyw Allium telomere był wskazówką w poszukiwaniu prawdziwego TR w roślinach lądowych. Nowo opisane TR w roślinach i dalsze badania telomerów / telomerazy w bazalnych kladach glonów mogą ujawnić cenne informacje na temat wczesnej ewolucji, dlatego badania telomerów roślinnych mogą znacząco przyczynić się do hipotez dotyczących pojawienia się eukariotów.
wkład autora
VP i SG w równym stopniu przyczyniły się do napisania, edycji i przygotowania tej mini-recenzji.,
finansowanie
konflikt interesów
autorzy oświadczają, że badanie zostało przeprowadzone w przypadku braku jakichkolwiek relacji handlowych lub finansowych, które mogłyby być interpretowane jako potencjalny konflikt interesów.
podziękowania
One Thousand Plant Transcriptomes Initiative (2019). Tysiąc transkryptów roślin i filogenomika roślin zielonych. Przyroda 574 (7780), 679-685. podoba mi się! do obserwowanych nr: 41586-019-1693-2
PubMed Abstract | CrossRef Full Text | Google Scholar
