Wie Sie in Abbildung 1 sehen können, variieren die Nukleotide nur geringfügig und nur in der stickstoffhaltigen Base. Im Falle von DNA sind diese Basen Adenin, Guanin, Cytosin und Thymin. Beachten Sie die Ähnlichkeit der Formen von Adenin und Guanin sowie die Ähnlichkeit zwischen Cytosin und Thymin. A und G werden als Purine klassifiziert, während C und T als Pyrimidine klassifiziert werden. Solange wir Dinge benennen, beachten Sie „Desoxyribose“und “ Ribose“. Wie der Name schon sagt, ist Desoxyribose nur eine Ribose ohne Sauerstoff., Genauer gesagt, wo eine Hydroxylgruppe an den 2-Kohlenstoff von Ribose gebunden ist, ist nur ein Wasserstoff an den 2-Kohlenstoff von Desoxyribose gebunden. Das ist der einzige Unterschied zwischen den beiden Zuckern.
Bei der zufälligen Konstruktion eines einzelnen Nukleinsäurestrangs in vitro gibt es keine besonderen Regeln für die Reihenfolge der Nukleotide in Bezug auf ihre Basen. Die Identitäten ihrer stickstoffhaltigen Basen sind irrelevant, da die Nukleotide durch Phosphodiesterbindungen durch die Phosphatgruppe und die Pentose gebunden sind., Es wird daher oft als Zucker-Phosphat-Rückgrat bezeichnet. Wenn wir das Wort „Phosphodiester“ aufschlüsseln, sehen wir, dass es die Verbindung recht handlich beschreibt: Die Zucker sind durch zwei Esterbindungen ( —O—) mit einem Phosphor dazwischen verbunden. Eine der Ideen, die Studenten oft verwirrt, ist die Direktionalität dieser Bindung und damit der Nukleinsäuren im Allgemeinen. Wenn wir zum Beispiel über DNA-Polymerase sprechen, das Enzym, das die Zugabe von Nukleotiden in lebenden Zellen katalysiert, sagen wir, dass es in einer Richtung von 5-Primzahl (5′) bis 3-Primzahl (3′) wirkt., Dies mag wie arkane Molekularbiologen sprechen, aber es ist eigentlich sehr einfach. Schauen Sie sich noch zwei der Nukleotide an, die durch die Phosphodiesterbindung miteinander verbunden sind (Abbildung \(\pageIndex{1}\), unten links). Ein Adenin-Nukleotid ist mit einem Cytosin-Nukleotid verbunden. Die Phosphodiesterbindung verbindet immer den 5-Kohlenstoff einer Desoxyribose (oder Ribose in RNA) mit dem 3-Kohlenstoff des nächsten Zuckers. Dies bedeutet auch, dass an einem Ende einer Kette von verbundenen Nukleotiden eine freie 5′ Phosphat (-PO4)- Gruppe und am anderen Ende ein freies 3′ Hydroxyl (- OH) vorhanden ist., Diese definieren die Direktionalität eines DNA-oder RNA-Strangs.

DNA wird normalerweise als doppelsträngiges Molekül in der Zelle gefunden, während RNA meist einzelsträngig ist., Es ist jedoch wichtig zu verstehen, dass unter den entsprechenden Bedingungen DNA einzelsträngig und RNA doppelsträngig gemacht werden kann. Tatsächlich sind die Moleküle so ähnlich, dass es sogar möglich ist, doppelsträngige Hybridmoleküle mit einem DNA-und einem RNA-Strang zu erzeugen. Interessanterweise sind RNA-RNA-Doppelhelix und RNA-DNA-Doppelhelix tatsächlich etwas stabiler als die konventionellere DNA-DNA-Doppelhelix.,
Die Basis der doppelsträngigen Natur der DNA und in der Tat die Basis von Nukleinsäuren als Medium für die Speicherung und Übertragung genetischer Informationen ist die Basenpaarung. Basenpaarung bezieht sich auf die Bildung von Wasserstoffbrücken zwischen Adeninen und Thyminen sowie zwischen Guaninen und Zytosinen. Diese Paare sind signifikant stabiler als jede Assoziation, die mit den anderen möglichen Basen gebildet wird. Wenn sich diese Basenpaarassoziationen weiterhin im Zusammenhang mit zwei Nukleinsäuresträngen bilden, ist ihr Abstand ebenfalls gleichmäßig und hochstabil., Sie erinnern sich vielleicht daran, dass Wasserstoffbrücken relativ schwache Bindungen sind. Im Kontext der DNA macht die Wasserstoffbindung die DNA jedoch extrem stabil und eignet sich daher gut als Langzeitspeichermedium für genetische Informationen. Da DNA – Doppelhelizen selbst bei einfachen Prokaryoten mindestens Tausende von Nukleotiden lang sind, bedeutet dies, dass mehrere tausend Wasserstoffbrücken die beiden Stränge zusammenhalten., Obwohl jede einzelne Nukleotid-zu-Nukleotid-Wasserstoffbindungs-Wechselwirkung leicht vorübergehend durch einen leichten Temperaturanstieg oder eine winzige Änderung der Ionenstärke der Lösung gestört werden kann, erfordert eine vollständige Doppelhelix von DNA sehr hohe Temperaturen (im Allgemeinen über 90 ° C), um die Doppelhelix vollständig in einzelne Stränge zu denaturieren.
Da es eine exakte Eins-zu-Eins-Paarung von Nukleotiden gibt, stellt sich heraus, dass die beiden Stränge im Wesentlichen Sicherungskopien voneinander sind – ein Sicherheitsnetz für den Fall, dass Nukleotide von einem Strang verloren gehen., Selbst wenn Teile beider Stränge beschädigt sind, solange der andere Strang im Bereich der Beschädigung intakt ist, ist die wesentliche Information immer noch in der komplementären Reihenfolge des gegenüberliegenden Strangs vorhanden und kann an Ort und Stelle geschrieben werden. Denken Sie jedoch daran, dass ein DNA – Strang zwar als „Backup“ des anderen fungieren kann, die beiden Stränge jedoch nicht identisch sind-sie ergänzen sich. Eine interessante Konsequenz dieses Systems komplementärer und antiparallelischer Stränge ist, dass die beiden Stränge jeweils einzigartige Informationen enthalten können.,
Bidirektionale Genpaare sind zwei Gene auf gegenüberliegenden DNA-Strängen, die jedoch einen Promotor teilen, der zwischen ihnen liegt. Da DNA nur in einer Richtung hergestellt werden kann, 5′ bis 3′, sendet dieser bidirektionale Promotor, oft eine CpG-Insel (siehe nächstes Kapitel), somit die RNA-Polymerase für jedes Gen in entgegengesetzte physikalische Richtungen. Dies wurde für eine Reihe von Genen gezeigt, die an Krebs (Brust, Eierstock) beteiligt sind, und ist ein Mechanismus zur Koordinierung der Expression von Netzwerken von Genprodukten.
Die Stränge einer DNA-Doppelhelix sind antiparallel., Dies bedeutet, dass, wenn wir eine Doppelhelix von DNA von links nach rechts betrachten, ein Strang in der 5′ bis 3′ Richtung konstruiert wäre, während der komplementäre Strang in der 3′ bis 5′ Richtung konstruiert ist. Dies ist wichtig für die Funktion von Enzymen, die DNA erzeugen und reparieren, wie wir bald diskutieren werden. In Abbildung \(\pageIndex{1}\) ist der linke Strang von oben nach unten 5′ bis 3′ und der andere von unten nach oben 5′ bis 3′.
Aus physikalischer Sicht sind DNA-Moleküle negativ geladen (all diese Phosphate) und normalerweise eine Doppelhelix mit einer rechtshändigen Drehung., In diesem normalen Zustand (auch als „B“ – Konformation bezeichnet) umfasst eine vollständige Drehung des Moleküls 11 Basenpaare mit 0,34 nm zwischen jeder Nukleotidbasis. Jede der stickstoffhaltigen Basen ist planar und bildet, wenn sie mit der komplementären Base gepaart wird, eine kleine planare „Sprosse“ auf der „Leiter“ der DNA. Diese sind senkrecht zur Längsachse der DNA. Der größte Teil der frei schwebenden DNA in einer Zelle und die meisten DNA in einer wässrigen Lösung von nahezu physiologischer Osmolarität und pH-Wert wird in dieser B-Konformation gefunden., Es wurden jedoch andere Konformationen gefunden, normalerweise unter sehr spezifischen Umweltbedingungen. Eine komprimierte Konformation, A-DNA, wurde als Artefakt der In-vitro-Kristallisation beobachtet, mit etwas mehr Basen pro Umdrehung, kürzerer Drehlänge und Basenpaaren, die nicht senkrecht zur Längsachse stehen. Eine andere, Z-DNA, scheint sich vorübergehend in GC-reichen DNA-Abschnitten zu bilden, in denen sich interessanterweise die DNA in die entgegengesetzte Richtung dreht.
Es wurde vorgeschlagen, dass sowohl die A-als auch die Z-Form der DNA physiologisch relevant sind., Es gibt Hinweise darauf, dass die A-Form in RNA-DNA-Hybrid-Doppelhelizen sowie auftreten kann, wenn DNA zu einigen Enzymen komplexiert ist. Die Z-Konformation kann als Reaktion auf die Methylierung der DNA auftreten. Darüber hinaus ist die“ normale “ B-DNA-Konformation eine idealisierte Struktur, die darauf basiert, vollständig hydratisiert zu sein, wie dies sicherlich sehr wahrscheinlich in einer Zelle der Fall ist. Dieser Hydratationszustand ändert sich jedoch ständig, wenn auch minutiös, so dass die DNA-Konformation oft geringfügig von den B-Konformationsparametern in Abbildung \(\pageIndex{2}\) variiert.,
In Prokaryoten wird die DNA im Zytoplasma gefunden (ziemlich offensichtlich, da es in diesen einfachen Organismen keine andere Wahl gibt), während in Eukaryoten die DNA im Kern gefunden wird. Trotz der Unterschiede in ihren Standorten, dem Schutzniveau vor äußeren Kräften und vor allem ihrer Größe ist sowohl prokaryotische als auch eukaryotische DNA mit Proteinen verpackt, die helfen, die gesamte Chromosomenstruktur zu organisieren und zu stabilisieren., Relativ wenig wird in Bezug auf die prokaryotische Chromosomenverpackung verstanden, obwohl strukturelle Ähnlichkeiten zwischen einigen der in prokaryotischen und eukaryotischen Chromosomen vorkommenden Proteine bestehen. Daher halten sich die meisten Einführungskurse für Zellbiologie an eukaryotische Chromosomenverpackungen.

Nackte DNA, ob prokaryotisch oder eukaryotisch, ist ein extrem dünner Materialstrang mit einem Durchmesser von etwa 11 nm. Angesichts der Größe der eukaryotischen Genome würde sich die DNA jedoch unkontrolliert verheddern, wenn sie auf diese Weise im Kern gespeichert würde. Stellen Sie sich einen Eimer vor, in den Sie hundert Meter Garn geworfen haben, ohne zu versuchen, es durch Aufwickeln oder Bündeln zu organisieren., Überlegen Sie nun, ob Sie in diesen Eimer greifen und an einem Strang ziehen können, und erwarten Sie, nur einen Strang hochzuziehen, oder ob Sie stattdessen wahrscheinlich mindestens ein kleines Gewirr Garn hochziehen. Die Zelle macht im Wesentlichen das, was Sie mit dem Garn tun würden, um es organisiert zu halten: Es ist ordentlich in kleinere, überschaubare Stränge verpackt. Im Falle der DNA wird jedes Chromosom um einen Histonkomplex geschlungen, um die erste Ordnung der Chromosomenorganisation zu bilden: das Nukleosom.
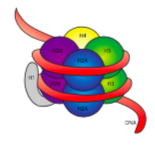
Die 30-nm-Faser wird durch zwei Wechselwirkungen zusammengehalten. Zunächst bringt das Linker-Histon H1 die Nukleosomen zu einer ungefähren 30-nm-Struktur zusammen., Diese Struktur wird dann durch Disulfidbindungen stabilisiert, die sich zwischen dem H2A-Histon eines Nukleosoms und dem H4-Histon seines Nachbarn bilden.
Histone sind eine Familie von basischen (positiv geladenen) Proteinen. Sie alle funktionieren hauptsächlich bei der Organisation von DNA, und das Nukleosom wird gebildet, wenn sich DNA (etwas mehr als zweimal) um einen Kern von acht Histonen wickelt – jeweils zwei von H2A, H2B, H3 und H4. Die Anzahl und Position der positiven Ladungen (meist aus Lysinen und Argininen) sind entscheidend für ihre Fähigkeit, DNA fest zu binden, die, wie bereits erwähnt, sehr negativ geladen ist., Diese“ Gegensätze ziehen “ Idee ist nicht nur ein Dating Tipp aus den Ratespalten.

Bei der Untersuchung der 3D-Struktur des Histonkernkomplexes sehen wir, dass, während relativ ungeladene Proteininteraktionsdomänen die Histone in der Mitte zusammenhalten, die positiv geladenen Reste um die Außenseite des Komplexes herum gefunden werden, die zur Interaktion mit den negativ geladenen Phosphaten der DNA zur Verfügung stehen.,
In einem späteren Kapitel werden wir diskutieren, wie Enzyme die DNA lesen, um ihre Informationen auf kleinere, besser handhabbare RNA-Teile zu transkribieren. Im Moment müssen wir uns nur bewusst sein, dass zu einem bestimmten Zeitpunkt ein Großteil der DNA dicht verpackt ist, während einige Teile der DNA dies nicht sind. Da die zur Verwendung verfügbaren Teile je nachdem, was zu einem bestimmten Zeitpunkt mit/in der Zelle passiert, variieren können, muss die Verpackung der DNA dynamisch sein. Es muss einen Mechanismus geben, um die Bindung von DNA an Histone schnell zu lockern, wenn diese DNA für die Genexpression benötigt wird, und die Bindung zu straffen, wenn dies nicht der Fall ist., Wie sich herausstellt, beinhaltet dieser Prozess Acetylierung und Deacetylierung der Histone.

Histon Acetyltransferasen (HATs) sind Enzyme, die eine Acetylgruppe auf ein Lysin eines Histonproteins setzen. Die Acetylgruppen sind negativ geladen, und die Acetylierung fügt nicht nur eine negativ geladene Gruppe hinzu, sondern entfernt auch die positive Ladung aus dem Lysin. Dies hat den Effekt, dass nicht nur ein Anziehungspunkt zwischen dem Protein und der DNA neutralisiert, sondern sogar leicht abgestoßen wird (mit ähnlichen Ladungen)., Auf der anderen Seite des Mechanismus sind Histon-Deactylasen (HDACs) Enzyme, die die Acetylierung entfernen und dadurch die Wechselwirkung zwischen Histon-Protein und DNA wiederherstellen. Da es sich um so wichtige Enzyme handelt, liegt es nahe, dass sie nicht willkürlich mit einem verfügbaren Histon arbeiten dürfen, und tatsächlich befinden sie sich häufig in einem Komplex mit anderen Proteinen, die ihre Aktivierung steuern und mit anderen Prozessen wie der Aktivierung der Transkription koordinieren.
