Introducción
los telómeros son estructuras nucleoproteicas en los extremos de los cromosomas eucarióticos lineales. Resuelven dos grandes problemas finales al mismo tiempo. La primera trata sobre la protección del extremo cromosómico. Se estima que las células humanas normales deben reparar al menos 50 roturas endógenas de doble cadena (DSBs) por célula por ciclo celular (Vilenchik y Knudson, 2003)., Los telómeros distinguen los extremos cromosómicos naturales de los DSBs dañinos y previenen su reparación ectópica, por ejemplo, mediante fusiones de extremo a extremo de los cromosomas (vanSteensel y deLange, 1997). El segundo es el problema de la replicación final que trata con el mantenimiento de longitudes de telómeros adecuadas. Esto fue reconocido de forma independiente por dos investigadores (Watson, 1972; Olovnikov, 1973). Dado que las polimerasas de ADN replicativas dependientes de ADN no pueden completar la síntesis de ADN en los extremos de los cromosomas, la compensación por la pérdida de secuencia de telómeros replicativa debe provenir de una polimerasa de ADN dependiente de ARN., Esta enzima, llamada telomerasa, junto con la primera secuencia de minisatélites de telómeros, fue descubierta en el Tetrahymena ciliado (Blackburn y Gall, 1978; Greider y Blackburn, 1985). Sin embargo, este es solo un aspecto del mantenimiento de la longitud de los telómeros. La regulación epigenética de la homeostasis de la longitud de los telómeros, incluyendo la interacción de la repetición telomérica larga no codificante que contiene ARN y las vías de actividad de la exonucleasa, también han sido ampliamente estudiadas debido a su potencial terapéutico (Wellinger et al., 1996; Polotnianka et al., 1998; Pfeiffer y Lingner, 2012).,
la telomerasa, la enzima encargada de añadir secuencias de repetición de telómeros al extremo 3′ de los telómeros, es una enzima compleja conservada con numerosos componentes . En principio, solo dos componentes principales son esenciales para la actividad enzimática de la telomerasa, un componente proteico catalíticamente activo, llamado transcriptasa inversa de los telómeros (TERT), y un componente plantilla, formado por la subunidad del ARN de la telomerasa (TR). Mientras que TERT es evolutivamente bastante bien conservado, TR es muy variable, con longitudes que van desde ca. 150 TN (Tetrahymena) a más de 2.000 TN (hongos del género Neurospora)., Solo una región corta en toda la molécula TR sirve como plantilla para el ADN telómero recién sintetizado (Greider y Blackburn, 1985; Qi et al., 2013). Esta región en TR se forma generalmente por un motivo telómero completo seguido por uno parcial, este último sirviendo como una región de recocido para el ADN telómero existente. Aunque, en principio, solo se necesita un único nucleótido extra (como motivo parcial), generalmente se encuentra más de uno. Por ejemplo, dos nucleótidos adicionales forman el motivo de recocido en ratones o cinco en humanos (Blasco et al., 1995; Feng et al., 1995)., En las plantas, sin embargo, el tamaño de la región de la plantilla es variable, por ejemplo, dos en Arabidopsis thaliana, siete en Arabis sp. o seis en Nicotiana (Fajkus et al., 2019). Las otras regiones TR tienen funciones estructurales, regulatorias y proteicas interactivas . Véase también una representación esquemática de la telomerasa y su ciclo de actividad en la Figura 1.

Figura 1 Representación esquemática del ciclo de actividad de la telomerasa con la plantilla de telómeros Tipo Arabidopsis. TERT: transcriptasa inversa de los telómeros; TR: subunidad del ARN de la telomerasa., Figura basada en Sekhri (2014).
¿qué tan variables son las secuencias de telómeros?
Las secuencias de telómeros suelen ser minisatélites cortos dispuestos en tándem, normalmente siguiendo la fórmula (TxAyGz) n. La disposición de los minisatélites se origina en la forma en que la telomerasa sintetiza el ADN, en resumen, y en su mayoría motivos idénticos, uno por uno., Varias hipótesis consideran que tal disposición es importante porque promueve el reconocimiento de proteínas específicas de telómeros por homo y heterodímeros y por el potencial de formar cuadruplejos G que pueden estabilizar los extremos cromosómicos o servir como sustratos para proteínas específicas de telómeros (Spiegel et al., 2020; Tran et al., 2013). Las secuencias de telómeros están bien conservadas a través de la evolución, y grandes grupos de organismos utilizan el motivo de grupo típico de los telómeros para construir su ADN de telómeros., Un número cada vez mayor de estudios y grandes pruebas de detección han demostrado que todos los vertebrados probados y muchos metazoos basales usan TTAGGG (Meyne et al., 1989; Traut et al., 2007) mientras que Euarthropoda (artrópodos), incluyendo Hexapoda (insectos), tienen TTAGG (Frydrychova et al., 2004; Vitkova et al., 2005). Constantemente, numerosas excepciones se están acumulando con el tiempo, por ejemplo, (A (G) 1-8) en Dictyostelium (Emery y Weiner, 1981), TTAGGC en Ascaris lumbricoides (Nematoda) (Muller et al., 1991), TCAGG en Coleoptera (escarabajos) (Mravinac et al., 2011), TAGGG/TAAGG/TAAGGG en Giardia (diplomonads) (Uzlikova et al.,, 2017), o TTNNNNAGGG en el clado de Yarrowia (levaduras) (Cervenak et al., 2019). Además, los sistemas independientes de la telomerasa, en los que la secuencia de telómeros minisatélites se ha perdido y sustituido por repeticiones complejas, están representados, por ejemplo, por Diptera y Chironomidae (revisado en (Mason et al., 2016)). Para una revisión general de la secuencia de telómeros eucarióticos ver (Fajkus et al., 2005; Fulneckova et al., 2013).
La composición de los telómeros en las plantas es aún más diversa., Aquí usamos el término «plantas» en un sentido amplio, también conocido como Archaeplastida o kingdom Plantae sensu lato, y comprende Rhodophyta (algas rojas), Glaucophyta, el grado de algas Clorofitas y el grado de algas Estreptofitas (en conjunto conocido como algas verdes), y Embryophyta (plantas terrestres) (iniciativa de mil transcriptomas de plantas, 2019). La secuencia típica de plantas de telómeros es TTTAGGG, también llamada Arabidopsis-type (O simplemente, plant-type) ya que fue descubierta en Arabidopsis thaliana (Richards y Ausubel, 1988) y ahora en muchas otras especies en casi todos los órdenes de plantas., Aunque TTTAGGG sigue siendo el más frecuente, existe una variabilidad significativa en las secuencias de telómeros en los linajes de algas rojas y verdes. En cuanto a las algas rojas (Rhodophyta), la información de la secuencia de telómeros es en su mayoría inexistente o fragmentaria, aunque algunos candidatos de telómeros se han descubierto in silico, como Aatggggg para Cyanidioschyzon merolae (Nozaki et al., 2007), TTATT(T) AGGG para Galdieria sulphuraria (Fulneckova et al., 2013); TTAGGG se ha encontrado en lecturas genómicas de Porphyra umbilicalis (Fulneckova et al., 2013), pero se necesita más evidencia para confirmar su posición terminal en los cromosomas., La diversidad de telómeros en las algas verdes refleja tanto los cambios dinámicos como su carácter parafilético. Aunque tttaggg prevalece en Chlorophyta, como en los géneros Ostreococcus (Derelle et al., 2006) y Chlorella (Higashiyama et al., 1995), muchos otros motivos divergentes también se han detectado allí, como TTAGGG en el género Dunaliella y Stephanosphaeria (Fulneckova et al., 2012), y TTTTAGGG en Chlamydomonas (Petracek et al., 1990). En Streptophyta basal (Klebsormidiophyceae) se han descrito cambios progresivos en los motivos de TTTAGGG a TTTTAGGG y TTTTAGG., La presencia de TTAGGG en Rhodophyta y Glaucophyta lleva a la hipótesis de que este es el motivo ancestral en las plantas (Archaeplastida) (Fulneckova et al., 2013).
en cuanto a las plantas terrestres, una de las primeras pruebas realizadas mostró que la secuencia Tipo Arabidopsis era la más común y se conservaba principalmente a través de su filogenia (Cox et al., 1993; Fuchs et al., 1995), aunque algunos de estos autores ya habían detectado varias excepciones en la familia Amaryllidaceae (ex Alliaceae), en la que la secuencia Tipo Arabidopsis estaba ausente en varias especies., Más tarde, la primera secuencia de telómeros inusual para las plantas terrestres, el tipo vertebrado TTAGGG, se caracterizó en Aloe y en algunos otros Asparagales (Weiss y Scherthan, 2002; Puizina et al., 2003; Sykorova et al., 2003c). Se formuló una hipótesis sobre pérdidas y recuperaciones repetidas de la secuencia de telómeros TTTAGGG y ttaggg en Asparagales (Adams et al., 2001). Con el postrefinamiento del Orden Asparagales en la APGIII (Angiosperm Phylogeny Group 2009) (Bremer et al.,, 2009), se demostró que solo dos cambios evolutivos importantes en la composición de la secuencia de telómeros ocurrieron (en lugar de varias pérdidas y ganancias repetidas), en el siguiente orden: el PRIMERO en la familia Iridaceae, en el que ocurrió un cambio del tipo de planta TTTAGGG al tipo de vertebrado TTAGGG, seguido por las familias Xeronemataceae, Asphodelaceae y el núcleo Asparagales (incluyendo Amarillidaceae S. l y Asparagaceae S. l.,); y la segunda dentro de la subfamilia Allioideae (anteriormente tratada como una familia separada, Alliaceae) en la que surgió una secuencia de telómeros completamente nueva, CTCGGTTATGGG (Fajkus et al., 2016). Fuera de Asparagales, también se han detectado nuevas secuencias de telómeros en grupos de plantas terrestres tan dispares como (I) Solanaceae, en las que se describió la secuencia de telómeros de Cestrum elegans ttttttaggg (Sykorova et al., 2003a; Sykorova et al., 2003b; Peska et al., 2008; Peska et al.,, 2015) y (ii) Lentibulariaceae, donde el género Genlisea mostró una notable diversidad con algunas especies caracterizadas por repeticiones de telómeros Tipo Arabidopsis, mientras que otras exhibieron Variantes de secuencia entremezcladas TTCAGG y TTTCAGG (Tran et al., 2015).
a pesar de todas las excepciones de motivos de telómeros detectadas, la diversidad real en secuencias teloméricas en plantas terrestres es probablemente muy subestimada. Una publicación reciente (Vitales et al.,, 2017), en el que se realizó una selección de secuencias de telómeros de plantas terrestres, encontró que las secuencias de telómeros solo se conocían claramente para menos del 10% de las especies y el 40% de los géneros contenidos en la base de datos de ADNr de plantas (www.plantrdnadatabase.com), un recurso que proporciona información de citogenética molecular sobre plantas terrestres (Garcia et al., 2012). Un resumen de la distribución de la secuencia de telómeros en plantas, después de APG IV (the Angiosperm Phylogeny Group, 2016) (Byng et al., 2016), así como la filogenia vegetal más reciente (iniciativa de mil transcriptomas de plantas, 2019) se encuentra en la Figura 2.,
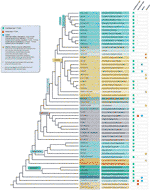
figura 2 motivos de telómeros en Archaplastida (plantas en sentido amplio), basados en la APG IV (the Angiosperm Phylogeny Group 2016) y en la iniciativa mil transcriptomas de plantas (2019). Las longitudes de rama no expresan escalas en tiempo real., Por simplicidad y para ahorrar espacio, ciertos «grupos» polifiléticos (grados) marcados con un asterisco en el árbol han sido representados por una sola rama; por la misma razón, varios órdenes menores (enumerados en el cuadrado azul en la parte superior izquierda de la figura) no se representan en el árbol. La primera etiqueta de punta generalmente se refiere a órdenes de plantas y, en algunos casos, a divisiones, grados e incluso familias; la segunda etiqueta muestra familias representativas y, en algunos casos, órdenes o géneros representativos.
de las proyecciones al descubrimiento: ¿cómo se pueden identificar los motivos Teloméricos?,
La evidencia de que una secuencia candidata dada es una telomérica Real incluye varios pasos que declaran correctamente su localización en todos los terminales cromosómicos, y finalmente la participación de la telomerasa en su síntesis. La citogenética Molecular (principalmente por hibridación fluorescente in situ, FISH) se ha vuelto importante para visualizar la localización terminal de sondas etiquetadas de secuencias candidatas en todos los terminales cromosómicos. Sin embargo, los peces independientes no es suficiente para demostrar la posición muy terminal., Por ejemplo, se propuso AcepSAT356 en cebolla como candidato a telómero, basándose en los resultados del análisis de FISH (Pich y Schubert, 1998). Sin embargo, su aparente localización terminal in situ nunca se ha relacionado de manera convincente con la función de los telómeros. En realidad, el descubrimiento de la secuencia de telómeros Allium minisatélite CTCGGTTATGGG y telomerasa significaría que AcepSAT356 es subterminal (Fajkus et al., 2019)., Las señales TELOMÉRICAS positivas de los peces también pueden enmascarar pequeños cambios en los motivos de los telómeros, como polimorfismos de un solo nucleótido, o los resultados falsos negativos pueden resultar de telómeros cortos que están por debajo del límite de detección de la técnica.
Hay dos enfoques adicionales que determinan la posición terminal a mayor resolución que FISH; estos se basan en la actividad de la exonucleasa BAL31., El primero es el análisis clásico de fragmentos de restricción Terminal (TRF), en el que las muestras tratadas con BAL31 muestran un acortamiento progresivo de los fragmentos terminales y una disminución de la intensidad de la señal con el aumento del tiempo de tratamiento con exonucleasas. El análisis posterior de las longitudes de los fragmentos se realiza mediante hibridación Southern-blot (Fojtova et al., 2015). El segundo es el desnatado comparativo del genoma (datos NGS) de ADN genómico no digerido y digerido por BAL31, en paralelo., En el conjunto de datos tratado BAL31, hay una infrarrepresentación significativa de las secuencias de telómeros, por lo tanto, las secuencias terminales se identifican en comparación con el conjunto de datos no tratado, utilizando herramientas bioinformáticas RepeatExplorer o Tandem Repeats Finder .
la otra prueba importante de un candidato de secuencia de telómeros dado en una especie es la demostración de la actividad de la telomerasa. En esto, un enfoque experimental útil, desarrollado primero para las células humanas, es el Protocolo de amplificación de repetición de telómeros (TRAP) (Kim et al., 1994), seguido de la secuenciación de los productos detectados (Peska et al.,, 2015; Fajkus et al., 2016), que es un poco menos sensible a los resultados falsos positivos que los peces. Todos estos métodos, incluidos los peces (Fuchs et al., 1995; Shibata y Hizume, 2011) y otros como la hibridación slot-blot (Sykorova et al., 2003C), y TRAP (Fulneckova et al., 2012; Fulneckova et al., 2016), se puede utilizar para detectar telómeros en grupos amplios de organismos complejos, incluidas las plantas., Sin embargo, solo una combinación de métodos adecuadamente elegidos puede conducir de manera convincente a una conclusión sobre la función telómera de una secuencia candidata, ya que los resultados basados en un solo enfoque pueden ser engañosos. Un resumen más completo de las estrategias para la identificación de novo de la secuencia de candidatos a telómeros, incluyendo el primer intento en Tetrahymena (Greider y Blackburn, 1985) se resumen en un artículo metodológico, con énfasis en el enfoque NGS utilizado en plantas con genomas extremadamente grandes (Peska et al., 2017).
¿hay Homoplasia en las secuencias de telómeros?,
se cree que la secuencia ancestral de telómeros es TTAGGG y es la más comúnmente encontrada en el árbol de la vida (Fulneckova et al., 2013). Sin embargo, parece claro que la frecuencia de homoplasia en la evolución de los motivos de los telómeros es relativamente alta. Por ejemplo, motivos cortos y simples como el tipo de planta TTTAGGG han aparecido de forma independiente y repetidamente en criptomonadas, hongos oomicetos y alveolatos; de manera similar, el tipo de vertebrado TTAGGG ha surgido de forma secundaria en ciertos grupos de plantas (Asparagales, Rodophyta y algas Clorofitas) (Sykorova et al., 2003C; Fulneckova et al.,, 2012; Fulneckova et al., 2013; Somanathan y Baysdorfer, 2018). La razón por la que algunas secuencias de telómeros han surgido con más frecuencia que otras, generalmente más complejas, está probablemente relacionada con las presiones de selección, lo que favorecería la precisión para una interacción ADN-proteína específica de una secuencia particular (Forstemann et al., 2003). Si hubiera un cambio en cada motivo de telómero, la interferencia en la estructura de la nucleoproteína telomérica conduciría necesariamente a la inestabilidad del genoma., Esta es la razón por la que las secuencias de telómeros son tan estables evolutivamente, comprendiendo muy pocas secuencias novedosas y exitosas, un patrón consistente con la idea de pérdidas repetidas y la aparición de las secuencias de telómeros típicas, como se propone para Asparagales (Adams et al., 2001).
el hallazgo de homoplasia a través de secuencias de telómeros plantea la pregunta, ¿Cuáles son las causas moleculares y los procesos que tienen lugar durante estos cambios? Un cambio en la secuencia de los telómeros, a pesar de parecer trivial en algunos casos (por ejemplo,,, una T extra), puede causar una interferencia grave con la integridad del genoma, debido a un equilibrio alterado en las interacciones entre el ADN y la proteína de los telómeros. Tampoco está claro si un cambio en la secuencia de los telómeros puede tener alguna ventaja evolutiva; en este sentido, (Tran et al., 2015) sugirió que la aparición de una citosina «metilable» en una cadena de telómeros ricos en G aumentaría la posibilidad de regulación por modificación epigenética.
¿Cuáles son las razones moleculares de los cambios en los motivos de los telómeros?,
para explicar el cambio de secuencia de telómeros, el primer candidato es la subunidad plantilla de la telomerasa, ARN de la telomerasa (TR). El TR previamente identificado de levaduras y vertebrados pertenece a un grupo diferente de transcripciones, cuya característica de conexión fue que fueron transcritas por ARN polimerasa II (Pol II)—en todos menos ciliados; esto solía ser la única excepción de las transcripciones Pol II antes de la publicación de la identificación de tr Planta terrestre . Usando el motivo relativamente largo del telómero de Allium para buscar su TR dentro del conjunto de datos de la secuencia total de ARN, Fajkus et al., (2019) mostraron que un ARN no codificante previamente caracterizado involucrado en la reacción de estrés en A. thaliana, llamado AtR8, era de hecho la subunidad de ARN telomerasa (Wu et al., 2012; Fajkus et al., 2019). Era una transcripción de la ARN polimerasa III (Pol III) que contenía los elementos reguladores correspondientes en su estructura promotora. Durante mucho tiempo, los investigadores esperaban que el TR de la planta fuera tan divergente que sería imposible identificarlo con base en una búsqueda de homología (Cifuentes-Rojas et al., 2011)., Sin embargo, un cierto grado de similitud se utilizó con éxito para identificar un TR común en varias especies de Allium con Blast comparativo. Sorprendentemente, la homología de la secuencia, la presencia de los mismos elementos reguladores y una región de plantilla correspondiente llevaron a la identificación de TRs en Allium, Arabidopsis y más de 70 otras plantas distantes, incluidas aquellas con motivos de telómeros divergentes como Genlisea, Cestrum y Tulbaghia., Hasta donde sabemos, todavía no hay datos sobre cualquier TR de algas, lo que aclararía si la transcripción de Pol III DE TR es una característica general para todas las plantas o no. Este trabajo (Fajkus et al., 2019), basado en CRISPR knock-out y otros experimentos, también mostró que un candidato de ARN telomerasa previamente identificado en A. thaliana (Cifuentes-Rojas et al., 2011; Beilstein et al., 2012)no fue una subunidad plantilla funcional de la telomerasa, como también fue demostrado poco después por (Dew-Budd et al., 2019)., Suponiendo que la dependencia de Pol II/Pol III para la transcripción de TR es un marcador evolutivo confiable, la futura investigación de TR en otros linajes eucariotas principales probablemente abrirá nuevas perspectivas sobre el origen de los eucariotas. Los genes de telomerasa y las secuencias de telómeros son fuentes no reconocidas de información en esta dirección, y el hallazgo de una vía de Biogénesis TR dependiente de Pol III en linajes de ciliados y plantas puede representar los primeros pasos en esta dirección (Greider y Blackburn, 1989; Fajkus et al., 2019).
¿cómo los cromosomas se volvieron lineales?,
una gran mayoría de procariotas contienen cromosomas circulares, mientras que los cromosomas lineales son la regla en los eucariotas., Por lo tanto, hay dos escenarios posibles en los que (i) la linealización se realizó por una telomerasa primitiva, precediendo a otros procesos que condujeron a las características y funciones cromosómicas lineales actuales o (ii) la linealización de un cromosoma circular prequariótico fue inicialmente independiente de la telomerasa, pero justo antes de que los eucariotas actuales divergieran, una telomerasa primitiva comenzó a ocupar los extremos de los cromosomas y se volvió esencial para los cromosomas lineales recién formados (Nosek et al., 2006). Villasante et al., (2007) propusieron un escenario evolutivo en el que la rotura del cromosoma circular procariótico ancestral activaba un mecanismo de transposición en los extremos del ADN, permitiendo la formación de telómeros por un mecanismo de replicación dependiente de la recombinación: las consecuencias de esta hipótesis llevaron a la sorprendente conclusión de que los centrómeros eucarióticos se derivaban de los telómeros.
curiosamente, el proceso opuesto a la linealización, es decir, la formación de cromosomas circulares (también llamados cromosomas de anillo) ha surgido de vez en cuando durante la evolución de los eucariotas, aunque es altamente inestable., Por ejemplo, en el caso de Amaranthus tuberculatus, los cromosomas de anillo aparecieron como una respuesta inducida por estrés, portando resistencia contra un herbicida (glifosato); estos cromosomas de anillo adicionales no mostraron hibridación con sondas de telómeros en el análisis de cariotipo (Koo et al., 2018). El sistema casi universal de la telomerasa y la excepcionalidad de los cromosomas circulares en los eucariotas no nos permiten apoyar una hipótesis sobre la otra., Sin embargo, la maquinaria recombinacional utilizada en el alargamiento alternativo de los telómeros (ALT), una vía independiente de la telomerasa, se asoció con ciertos cánceres humanos (Zhang et al., 2019), ya está presente en prokaryotes. Además, hay evidencia de linealización cromosómica que ocurre independientemente en distintos linajes procariotas (Ferdows y Barbour, 1989; Nosek et al., 1995; Volff y Altenbuchner, 2000). Por lo tanto, la hipótesis de que el primer cromosoma eucariótico lineal (originario de un antepasado procariota) era independiente de la telomerasa parece más probable., Hay algunos ejemplos que muestran que el sistema basado en la telomerasa no es esencial para el mantenimiento de los telómeros en todos los eucariotas: retrotransposones en los telómeros de Drosophila, repeticiones satelitales en Quironomus, otro insecto (Rubin, 1978; Biessmann y Mason, 2003), y ALT en cánceres humanos con telomerasa negativa (Hu et al., 2016; Zhang et al., 2019). Sin embargo, algunos de estos sistemas pueden no ser tan diferentes, y quizás compartan un origen común: en Drosophila, el mantenimiento de los telómeros, basado en la retrotransposición, no es demasiado distinto del mecanismo basado en la telomerasa (Danilevskaya et al.,, 1998), lo que lleva a la hipótesis de que la propia telomerasa puede ser un antiguo retrotransposón. Pero ciertamente, las especies de plantas negativas a la telomerasa no se han descubierto hasta la fecha y todas las excepciones, en las que el típico telómero de tipo planta estaba ausente, se demostró más tarde que tenían motivos diferentes, pero aún sintetizados por la telomerasa. Sin embargo, la maquinaria ALT está presente en plantas en paralelo a la actividad telomerasa (Watson y Shippen, 2007; Ruckova et al., 2008). Preguntas interesantes sobre el papel de la telomerasa, los telómeros y su mantenimiento en los tumores de plantas surgen de eso., Uno atractivo es la ausencia de metástasis en las plantas, a pesar de la presencia de ALT, tal vez relacionada con la rigidez del tejido vegetal o sistemas inmunológicos diferentes que en los animales (Seyfried y Huysentruyt, 2013).
aunque estamos adquiriendo un conocimiento cada vez mayor de la biología de los telómeros, todavía somos incapaces de explicar la aparición de la telomerasa en los eucariotas., La evidencia actual apoya la hipótesis de que la aparición de eucariotas junto con sus cromosomas lineales, telómeros y telomerasa se relacionó con la aparición de intrones spliceosómicos en huéspedes arqueales (Koonin, 2006; Fajkus et al., 2019). La similitud entre TERT y otros retroelementos ha sido discutida por algún tiempo (Pardue et al., 1997). Sorprendentemente, un estudio relativamente reciente mostró que TERT, como probable miembro de la progenie de intrones del Grupo II, está secuencialmente cerca de los retrotransposones de elementos similares a Penélope (Gladyshev y Arkhipova, 2007)., Pero TERT es solo uno de los dos componentes esenciales de la telomerasa, y TR es, en su origen, aún más enigmático debido a su baja conservación de secuencias en todos los eucariotas .
conclusión
al inicio de la era de la genómica de las plantas, la secuencia de telómeros se consideraba casi invariable. La conservación general de los telómeros y el sistema de telomerasa sugiere que todas las plantas pueden tener el telómero tipo planta TTTAGGG. La identificación de secuencias de telómeros inusuales en genomas de plantas complejas, en muchos casos con valores C gigantes(como en Cestrum y Allium sp.,), valió la pena el esfuerzo, ya que el excepcionalmente largo motivo de Allium telomere fue la clave para buscar un TR genuino en las plantas terrestres. El TR recientemente descrito en plantas y la investigación posterior de telómeros/telomerasas en clados basales de algas podrían revelar información valiosa sobre la evolución temprana, por lo tanto la investigación de telómeros de plantas puede contribuir significativamente a las hipótesis sobre la aparición de eucariotas.
contribuciones del autor
VP y SG han contribuido igualmente a la redacción, edición y preparación de esta mini-revisión.,
financiación
conflicto de intereses
los autores declaran que la investigación se realizó en ausencia de relaciones comerciales o financieras que pudieran ser interpretadas como un potencial conflicto de intereses.
agradecimientos
iniciativa de transcriptomas de mil plantas (2019). One thousand plant transcriptomes and the phylogenomics of green plants (en inglés). Nature 574 (7780), 679-685. doi: 10.1038 / s41586-019-1693-2
resumen de PubMed / texto completo de CrossRef / Google Scholar
