Einführung
Telomere sind Nukleoproteinstrukturen an den Enden linearer eukaryotischer Chromosomen. Sie lösen zwei große Endprobleme gleichzeitig. Die erste ist über Chromosomenendschutz. Es wird geschätzt, dass normale menschliche Zellen mindestens 50 endogene doppelsträngige Brüche (DSBs) pro Zelle und Zellzyklus reparieren müssen (Vilenchik und Knudson, 2003)., Telomere unterscheiden die natürlichen Chromosomenenden von schädlichen DSBs und verhindern deren ektopische Reparatur, z. B. durch End-to-End-Fusionen von Chromosomen (vanSteensel und DeLange, 1997). Das zweite ist das Endreplikationsproblem, das sich mit der Aufrechterhaltung der richtigen Telomerlängen befasst. Dies wurde unabhängig von zwei Forschern erkannt (Watson, 1972; Olovnikov, 1973). Da replikative DNA-abhängige DNA-Polymerasen die DNA-Synthese an den Enden der Chromosomen nicht vollständig abschließen können, muss die Kompensation für den Verlust replikativer Telomersequenz von einer RNA-abhängigen DNA-Polymerase stammen., Dieses Enzym, Telomerase genannt, wurde zusammen mit der ersten Telomerminisatellitensequenz in der Ciliatentetrahymena entdeckt (Blackburn und Gall, 1978; Greider und Blackburn, 1985). Dies ist jedoch nur ein Aspekt der Wartung der Telomerlänge. Die epigenetische Regulation der telomerlangen Homöostase, einschließlich der Wechselwirkung von langen, nicht codierenden, RNA-haltigen telomeren Zellen und Exonukleaseaktivitätswegen, wurde aufgrund ihres therapeutischen Potenzials ebenfalls umfassend untersucht (Wellinger et al., 1996; Polotnianka et al., 1998; Pfeiffer und Lingner, 2012).,
Telomerase, das Enzym, das für die Zugabe von Telomer-Wiederholungssequenzen zum 3′ – Ende von Telomeren verantwortlich ist, ist ein konserviertes komplexes Enzym mit zahlreichen Komponenten . Grundsätzlich sind nur zwei Hauptkomponenten für die enzymatische Telomerase-Aktivität essentiell, eine katalytisch aktive Proteinkomponente, die Telomerase-Reverse-Transkriptase (TERT) genannt wird, und eine Template-Komponente, die von der Telomerase-RNA-Untereinheit (TR) gebildet wird. Während TERT evolutionär recht gut konserviert ist, ist TR sehr variabel,mit Längen von ca. 150 nt (Tetrahymena) bis über 2.000 nt (Pilze der Gattung Neurospora)., Nur eine kurze Region im gesamten TR-Molekül dient als Vorlage für neu synthetisierte Telomerdna (Greider und Blackburn, 1985; Qi et al., 2013). Dieser Bereich in TR wird üblicherweise durch ein vollständiges Telomermotiv gebildet, gefolgt von einem partiellen, wobei letzterer als Glühbereich für die vorhandene Telomerdna dient. Obwohl im Prinzip nur ein einzelnes zusätzliches Nukleotid benötigt wird (als Teilmotiv), wird normalerweise mehr als eines gefunden. Zum Beispiel bilden zwei zusätzliche Nukleotide das Glühmotiv bei Mäusen oder fünf beim Menschen (Blasco et al., 1995; Feng et al., 1995)., Bei Pflanzen ist die Größe der Vorlagenregion jedoch variabel, z. B. zwei in Arabidopsis thaliana, sieben in Arabis sp. oder sechs in Nicotiana (Fajkus et al., 2019). Die anderen TR-Regionen haben strukturelle, regulatorische und proteinbasierte Funktionen . Siehe auch eine schematische Darstellung der Telomerase und ihres Aktivitätszyklus in Abbildung 1.

Abbildung 1 Schematische Darstellung des Telomerase-Aktivitätszyklus mit der Telomervorlage vom Typ Arabidopsis. TERT, Telomere Reverse Transkriptase; TR, Telomerase-RNA-Untereinheit., Abbildung basierend auf Sekhri (2014).
Wie Variabel Sind die Telomer-Sequenzen?
Telomersequenzen sind in der Regel kurze Minisatelliten tandemartig angeordnet, typischerweise nach der Formel (TxAyGz) n. Die Minisatellitenanordnung stammt aus der Art und Weise, wie Telomerase die DNA synthetisiert, kurz und meist identische Motive nacheinander., Mehrere Hypothesen halten eine solche Anordnung für wichtig, da sie die Erkennung telomerspezifischer Proteine durch Homo – und Heterodimer fördert und das Potenzial hat, G-Quadruplexe zu bilden, die Chromosomenenden stabilisieren oder als Substrate für telomerspezifische Proteine dienen können (Spiegel et al., 2020; Tran et al., 2013). Telomersequenzen sind durch Evolution gut konserviert, und große Gruppen von Organismen verwenden das gruppentypische Telomermotiv, um ihre Telomer-DNA aufzubauen., Eine allmählich zunehmende Anzahl von Studien und großen Screenings hat gezeigt, dass alle getesteten Wirbeltiere und viele basale Metazoane TTAGGG verwenden (Meyne et al., 1989; Traut et al., 2007) während Euarthropoden (Arthropoden), einschließlich Hexapoda (Insekten), TTAGG haben (Frydrychova et al., 2004; Vitkova et al., 2005). Stetig, zahlreiche Ausnahmen häufen im Laufe der Zeit, z.B., (A(G)1-8) in Dictyostelium (Emery und Weiner, 1981), TTAGGC in Ascaris lumbricoides (Nematoda) (Muller et al., 1991), TCAGG in Coleoptera (Käfer) (Mravinac et al., 2011), TAGGG / TAAGG / TAAGGG in Giardia (Diplomonaden) (Uzlikova et al.,, 2017), oder TTNNNNAGGG in Yarrowia clade (Hefen) (Cervenak et al., 2019). Darüber hinaus werden telomeraseunabhängige Systeme, bei denen die Minisatelliten-Telomersequenz verloren gegangen und durch komplexe Wiederholungen ersetzt wurde, beispielsweise durch Diptera und Chironomidae dargestellt (rezensiert in (Mason et al., 2016)). Eine allgemeine Übersicht über die eukaryotische Telomersequenz finden Sie unter (Fajkus et al., 2005; Fulneckova et al., 2013).
Die Telomerzusammensetzung in Pflanzen ist noch vielfältiger., Hier verwenden wir den Begriff „Pflanzen“ im weiteren Sinne, auch bekannt als Archaeplastida oder Kingdom Plantae sensu lato, und umfassen Rhodophyta (Rotalgen), Glaukophyta, die Chlorophytalgenqualität und die Streptophytalgenqualität (insgesamt bekannt als Grünalgen) und Embryophyta (Landpflanzen) (Eintausend Pflanzentranskriptome Initiative, 2019). Die typische Telomerpflanzensequenz ist TTTAGGG, auch Arabidopsis-Typ (oder einfach Pflanzenart) genannt, da sie in Arabidopsis thaliana (Richards und Ausubel, 1988) und jetzt in vielen anderen Arten in fast allen Pflanzenordnungen entdeckt wurde., Obwohl TTTAGGG immer noch am häufigsten vorkommt, besteht eine signifikante Variabilität der Telomersequenzen in roten und grünen Algenlinien. Bei Rotalgen (Rhodophyta) fehlen Telomersequenzinformationen meist oder sind fragmentarisch, obwohl einige Telomerkandidaten in Silico entdeckt wurden, wie AATGGGGGG für Cyanidioschyzon merolae (Nozaki et al., 2007), TTATT (T)AGGG für Galdieria sulfuraria (Fulneckova et al., 2013); TTAGGG wurde in genomischen Proben von Porphyra umbilicalis gefunden (Fulneckova et al., 2013), aber mehr Beweise sind erforderlich, um ihre Endposition auf Chromosomen zu bestätigen., Die Telomervielfalt in Grünalgen spiegelt sowohl dynamische Veränderungen als auch ihren paraphyletischen Charakter wider. Obwohl TTTAGGG in Chlorophyta vorherrscht, wie in den Gattungen Ostreococcus (Derelle et al. 2006) und Chlorella (Higashiyama et al., 1995) wurden auch dort viele andere divergierende Motive entdeckt, wie TTAGGG in der Gattung Dunaliella und Stephanosphaeria (Fulneckova et al., 2012), und TTTTAGGG in Chlamydomonas (Petracek et al., 1990). In basal Streptophyta (Klebsormidiophyceae) wurden progressive Veränderungen der Motive von TTTAGGG zu TTTTAGGG und TTTTAGG beschrieben., Das Vorhandensein von TTAGGG in Rhodophyta und Glaukophyta führt zu der Hypothese, dass dies das Ahnenmotiv in Pflanzen (Archaeplastida) ist (Fulneckova et al., 2013).
In Bezug auf Landpflanzen zeigte eines der ersten Screenings, dass die Arabidopsis-Typ-Sequenz am häufigsten war und größtenteils durch ihre Phylogenie konserviert wurde (Cox et al., 1993; Fuchs et al., 1995), obwohl einige dieser Autoren bereits einige Ausnahmen in der Familie Amaryllidaceae (ehemalige Alliaceae) festgestellt hatten, in der die Arabidopsis-Typsequenz bei mehreren Arten fehlte., Später wurde die erste für Landpflanzen ungewöhnliche Telomersequenz, die wirbeltierartige TTAGGG, in Aloe und in einigen anderen Asparagalen charakterisiert (Weiss und Scherthan, 2002; Puizina et al., 2003; Sykorova et al., 2003c). Eine Hypothese über wiederholte Verluste und Wiederherstellungen der TTTAGGG-und TTAGGG-Telomersequenz in Asparagales wurde formuliert (Adams et al., 2001). Mit der postrefinement der Ordnung Asparagales in der APGIII (Angiospermen Phylogenie Gruppe 2009) (Bremer et al.,, 2009), wurde gezeigt, dass nur zwei große evolutionäre Schalter in der Telomersequenz Zusammensetzung aufgetreten (anstatt mehrere wiederholte Verluste und Gewinne), in der folgenden Reihenfolge: die erste in der Familie Iridaceae, in denen eine Verschiebung von der Pflanze-Typ TTTAGGG zu den Wirbeltier-Typ TTAGGG passiert, gefolgt von Familien Xeronemataceae, Asphodelaceae und der Kern Asparagales (einschließlich Amarillidaceae s. l. und Asparagaceae s. l.,); und die zweite innerhalb der Unterfamilie Allioideae (früher als separate Familie behandelt, Alliaceae), in der eine völlig neue Telomersequenz entstand, CTCGGTTATGGG (Fajkus et al., 2016). Außerhalb von Asparagalen wurden auch neue Telomersequenzen in so unterschiedlichen Landpflanzengruppen wie (i) Solanaceae nachgewiesen, in denen die Telomersequenz von Cestrum elegans TTTTTTAGGG beschrieben wurde (Sykorova et al., 2003a; Sykorova et al., 2003b; Peska et al., 2008; Peska et al.,, 2015) und (ii) Lentibulariaceae, wo die Gattung Genlisea eine bemerkenswerte Vielfalt zeigte, wobei einige Arten durch die Telomerwiederholungen vom Arabidopsis-Typ gekennzeichnet waren, während andere vermischte Sequenzvarianten TTCAGG und TTTCAGG zeigten (Tran et al., 2015).
Trotz aller festgestellten Telomermotiv-Ausnahmen wird die wirkliche Vielfalt telomerer Sequenzen in Landpflanzen wohl stark unterschätzt. Eine aktuelle Publikation (Vitales et al.,, 2017), bei dem ein Screening von Landpflanzentelomersequenzen durchgeführt wurde, festgestellt, dass Telomersequenzen nur für weniger als 10% der Arten und 40% der in der Pflanzen-rDNA-Datenbank enthaltenen Gattungen eindeutig bekannt waren (www.plantrdnadatabase.com), a resource providing molecular cytogenetics information on land plants (Garcia et al., 2012). Eine Zusammenfassung der Telomer-Sequenz-Verteilung in Pflanzen nach APG IV (Die Angiospermen Phylogenie Gruppe, 2016) (Byng et al., 2016), sowie die jüngste Pflanzenphylogenie (Eintausend Pflanzentranskriptome Initiative, 2019) ist in Abbildung 2 zu finden.,
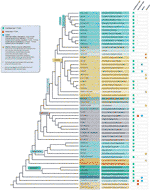
Abbildung 2 Telomermotive in Archaeplastida (Pflanzen im weiteren Sinne), basierend auf der APG IV (Angiosperm Phylogeny Group 2016) und der One Thousand Plant Transcriptomes Initiative (2019). Verzweigungslängen drücken keine Echtzeitskalen aus., Der Einfachheit halber und um Platz zu sparen, wurden bestimmte polyphyletische „Gruppen“ (Noten), die im Baum mit einem Sternchen markiert sind, durch einen einzigen Zweig dargestellt; Aus dem gleichen Grund sind mehrere Nebenordnungen (aufgeführt im blauen Quadrat an der linken oberen Seite der Figur) nicht auf dem Baum dargestellt. Das erste Spitzenetikett bezieht sich normalerweise auf Pflanzenbestellungen und in einigen Fällen auf Abteilungen, Sorten und sogar Familien; Das zweite Etikett zeigt repräsentative Familien und in einigen Fällen repräsentative Bestellungen oder Gattungen an.
Von Screenings zur Erkenntnis: Wie Telomeric Motive Können Erkannt Werden?,
Der Beweis, dass eine gegebene Kandidatensequenz eine echte telomere Sequenz ist, umfasst mehrere Schritte, die ihre Lokalisierung an allen chromosomalen Termini und schließlich die Beteiligung der Telomerase an ihrer Synthese richtig deklarieren. Die molekulare Zytogenetik (meist durch Fluoreszenz-in-situ-Hybridisierung, FISH) ist wichtig geworden, um die terminale Lokalisation markierter Sonden Kandidatensequenzen an allen chromosomalen Termini zu visualisieren. Für FISCHE reicht es jedoch nicht aus, die Endposition zu beweisen., Zum Beispiel wurde AcepSAT356 in Russland als Telomerkandidat vorgeschlagen, basierend auf Ergebnissen der Fischanalyse (Pich und Schubert, 1998). Nichtsdestotrotz wurde seine scheinbare Terminalposition durch In situ nie überzeugend mit der Telomerfunktion in Verbindung gebracht. Tatsächlich würde die Entdeckung der Allium-Minisatelliten-Telomersequenz CTCGGTTATGGG und Telomerase bedeuten, dass AcepSATase subterminal ist (Fajkus et al., 2019)., Positive telomere Fischsignale können auch winzige Veränderungen in Telomermotiven wie Einzelnukleotidpolymorphismen maskieren, oder falsch negative Ergebnisse können dazu führen, dass kurze Telomere unterhalb der Nachweisgrenze der Technik liegen.
Es gibt zwei zusätzliche Ansätze, die die Endposition bei größerer Auflösung als FISCH bestimmen; Diese basieren auf Exonuklease-B31-Aktivität., Die erste ist die klassische TRF-Analyse (Terminal Restriction Fragment), bei der mit BAL31 behandelte Proben eine fortschreitende Verkürzung der terminalen Fragmente und eine Abnahme der Signalintensität mit zunehmender Zeit der Exonuklease-Behandlung zeigen. Die anschließende Analyse der Fragmentlängen erfolgt durch Southern-Blot-Hybridisierung (Fojtova et al., 2015). Die zweite ist das vergleichende Genom-Skimming (NGS-Daten) von nicht verdauter und balsamverdauter genomischer DNA parallel., In dem BAL31 behandelten Datensatz gibt es eine signifikante Unterdarstellung von Telomersequenzen, daher werden die Terminalsequenzen durch Vergleich mit dem unbehandelten Datensatz mithilfe von Bioinformatikwerkzeugen RepeatExplorer oder Tandem Repeats Finder identifiziert .
Der andere wichtige Test eines gegebenen Telomersequenzkandidaten bei einer Spezies ist der Nachweis der Telomeraseaktivität. Ein nützlicher experimenteller Ansatz, der zuerst für menschliche Zellen entwickelt wurde, ist dabei das Telomere Repeat Amplification Protocol (TRAP) (Kim et al., 1994), gefolgt von der Sequenzierung der nachgewiesenen Produkte (Peska et al.,, 2015; Fajkus et al., 2016), die etwas weniger empfindlich auf falsch positive Ergebnisse reagiert als FISCHE. Alle diese Methoden, einschließlich FISCH (Fuchs et al., 1995; Shibata und Hizume, 2011) und andere wie Slot-Blot-Hybridisierung (Sykorova et al., 2003c) und TRAP (Fulneckova et al., 2012; Fulneckova et al., 2016), kann verwendet werden, um nach Telomeren in großen Gruppen komplexer Organismen, einschließlich Pflanzen, zu suchen., Allerdings kann nur eine Kombination von entsprechend gewählten Methoden überzeugend zu einer Schlussfolgerung über die Telomerfunktion einer Kandidatensequenz führen, da Ergebnisse, die auf einem einzigen Ansatz basieren, irreführend sein können. Ein vollständigerer Überblick über die Strategien zur Identifizierung von De novo-Telomerkandidaten, einschließlich des allerersten Versuchs an Tetrahymena (Greider und Blackburn, 1985), wird in einem methodischen Artikel zusammengefasst, wobei der Schwerpunkt auf dem NGS-Ansatz liegt, der in Pflanzen mit extrem großen Genomen verwendet wird (Peska et al., 2017).
Gibt es Homoplasie in Telomersequenzen?,
Die Vorfahren der Telomer-Sequenz ist gedacht, um ttaggg eingehen, und ist die am häufigsten über den Baum des Lebens (Fulneckova et al., 2013). Es scheint jedoch klar zu sein, dass die Häufigkeit der Homoplasie in der Telomerepithel-Evolution relativ hoch ist. Zum Beispiel sind kurze, einfache Motive wie der Pflanzentyp TTTAGGG unabhängig und wiederholt in Kryptomonaden, Oomyceten-Pilzen und Alveolen aufgetaucht; In ähnlicher Weise ist der Wirbeltiertyp TTAGGG sekundär in bestimmten Pflanzengruppen (Asparagales, Rodophyta und Chlorophyta-Algen) aufgetreten (Sykorova et al., 2003; Fulneckova et al.,, 2012; Fulneckova et al., 2013; Somanathan und Baysdorfer, 2018). Der Grund, warum einige Telomersequenzen häufiger aufgetreten sind als andere, meist komplexere Sequenzen, hängt wahrscheinlich mit dem Selektionsdruck zusammen, was die Genauigkeit für eine bestimmte sequenzspezifische DNA-Protein-Interaktion begünstigen würde (Forstemann et al., 2003). Bei einer Veränderung jedes Telomermotivs würde eine Interferenz in der telomeren Nukleoproteinstruktur notwendigerweise zu einer Genominstabilität führen., Dies ist der Grund, warum Telomersequenzen so evolutionär stabil sind und nur sehr wenige neuartige und erfolgreiche Sequenzen umfassen, ein Muster, das mit der Idee wiederholter Verluste und der Entstehung der typischen Telomersequenzen übereinstimmt, wie für Asparagales vorgeschlagen (Adams et al., 2001).
Der Befund der Homoplasie über Telomersequenzen hinweg wirft die Frage auf, welche molekularen Ursachen und Prozesse während dieser Verschiebungen stattfinden. Eine Änderung der Telomersequenz, obwohl sie in einigen Fällen trivial erscheint (z.,, ein zusätzliches T), kann aufgrund eines gestörten Gleichgewichts in den Telomer-DNA-Protein-Wechselwirkungen zu schwerwiegenden Störungen der Genomintegrität führen. Es ist auch unklar, ob eine Änderung der Telomersequenz evolutionären Vorteil haben kann; in dieser Hinsicht (Tran et al., 2015) vorgeschlagen, dass das Auftreten eines“ methylierbaren “ Cytosins in einem G-reichen Telomerstrang die Möglichkeit einer Regulation durch epigenetische Modifikation erhöhen würde.
Was sind die molekularen Gründe für Veränderungen der Telomermotive?,
Um die Veränderung der Telomersequenz zu erklären, ist der erste Kandidat die Template-Untereinheit der Telomerase, Telomerase RNA (TR). Das zuvor identifizierte TR aus Hefe und Wirbeltieren gehört zu einer anderen Gruppe von Transkripten, deren Verbindungsmerkmal darin bestand, dass sie durch RNA—Polymerase II (Pol II) transkribiert wurden-in allen außer Ciliaten; Dies war die einzige Ausnahme von Pol II-Transkripten vor Veröffentlichung der Land Plant TR-Identifizierung . Durch die Verwendung des relativ langen Telomermotivs von Allium, um nach seinem TR innerhalb des gesamten RNA-Sequenzdatenpools zu suchen, Fajkus et al., (2019) zeigte, dass eine zuvor charakterisierte nichtcodierende RNA, die an der Stressreaktion in A. thaliana beteiligt war, AtR8 genannt, tatsächlich die Telomerase-RNA-Untereinheit war (Wu et al., 2012; Fajkus et al., 2019). Es war ein Transkript der RNA-Polymerase III (Pol III), das die entsprechenden regulatorischen Elemente in seiner Promotorstruktur enthielt. Lange Zeit erwarteten die Forscher, dass die Pflanzenstruktur so unterschiedlich sein würde, dass es unmöglich wäre, sie anhand einer homologischen Suche zu identifizieren (Cifuentes-Rojas et al., 2011)., Ein gewisses Maß an Ähnlichkeit wurde jedoch erfolgreich verwendet, um eine gemeinsame TR in mehreren Allium-Arten mit vergleichenden Methoden zu identifizieren. Überraschenderweise führten die Sequenzhomologie, das Vorhandensein derselben regulatorischen Elemente und eine entsprechende Vorlagenregion zur Identifizierung von TRs in Allium, Arabidopsis und mehr als 70 anderen fern verwandten Pflanzen, einschließlich solcher mit divergierten Telomermotiven wie Genlisea, Cestrum und Tulbaghia., Soweit wir wissen, gibt es noch keine Daten zu Algen-TR, die Aufschluss darüber geben würden, ob die Pol III-Transkription von TR ein allgemeines Merkmal für alle Pflanzen ist oder nicht. Diese Arbeit (Fajkus et al., 2019), basierend auf CRISPR Knock-out und anderen Experimenten, zeigte auch, dass ein zuvor identifizierter Telomerase-RNA-Kandidat in A. thaliana (Cifuentes-Rojas et al., 2011; Beilstein et al., 2012) war keine funktionelle Template-Untereinheit der Telomerase, wie auch kurz danach gezeigt wurde (Dew-Budd et al., 2019)., Unter der Annahme, dass die Pol II/Pol III-Abhängigkeit für die TR-Transkription ein zuverlässiger evolutionärer Marker ist, werden zukünftige TR-Forschungen in anderen eukaryotischen Hauptlinien wahrscheinlich neue Erkenntnisse über den Ursprung von Eukaryoten eröffnen. Telomerase-Gene und Telomersequenzen sind in dieser Richtung unerkannte Informationsquellen, und das Auffinden eines Pol III-abhängigen TR-Biogeneseweges in Ciliaten-und Pflanzenlinien kann die ersten Schritte in diese Richtung darstellen (Greider und Blackburn, 1989; Fajkus et al., 2019).
Wie wurden Chromosomen Linear?,
Die überwiegende Mehrheit der Prokaryoten enthält kreisförmige Chromosomen, während lineare Chromosomen bei Eukaryoten die Regel sind., Daher gibt es zwei mögliche Szenarien, in denen entweder (i) die Linearisierung durch eine primitive Telomerase durchgeführt wurde, die anderen Prozessen vorausging, die zu aktuellen linearen chromosomalen Merkmalen und Funktionen führten, oder (ii) die Linearisierung eines prä-eukaryotischen zirkulären Chromosoms anfangs telomeraseunabhängig war, aber kurz bevor aktuelle Eukaryoten divergierten, begann eine primitive Telomerase Chromosomenenden zu besetzen und wurde für die neu gebildeten linearen Chromosomen essentiell (Nosek et al., 2006). Villasante et al., (2007) schlug ein evolutionäres Szenario vor, in dem der Bruch des prokaryotischen zirkulären Chromosoms der Vorfahren einen Transpositionsmechanismus an DNA-Enden aktivierte, der die Bildung von Telomeren durch einen rekombinationsabhängigen Replikationsmechanismus ermöglichte: Folgen dieser Hypothese führten zu der überraschenden Schlussfolgerung, dass eukaryotische Zentromer von Telomeren abgeleitet wurden.
Interessanterweise ist der entgegengesetzte Prozess der Linearisierung, d. H. Die Bildung zirkulärer Chromosomen (auch Ringchromosomen genannt), während der Evolution von Eukaryoten von Zeit zu Zeit aufgetreten, obwohl er sehr instabil ist., Zum Beispiel erschienen im Fall von Amaranthus tuberculatus Ringchromosomen als stressinduzierte Reaktion, die Resistenz gegen ein Herbizid (Glyphosat) tragend; Diese zusätzlichen Ringchromosomen zeigten keine Hybridisierung mit Telomersonden in der Karyotypanalyse (Koo et al., 2018). Das fast universelle Telomerasesystem und die Außergewöhnlichkeit zirkulärer Chromosomen in Eukaryoten erlauben es uns nicht, eine Hypothese über die andere zu stützen., Die bei der alternativen Verlängerung von Telomeren (ALT), einem Telomerase-unabhängigen Weg, verwendeten Rekombinationsmaschinen sind jedoch mit bestimmten menschlichen Krebsarten assoziiert (Zhang et al., 2019), ist bereits in Prokaryoten. Darüber hinaus gibt es Hinweise auf eine Chromosomenlinearisierung, die unabhängig in verschiedenen Prokaryotenlinien auftritt (Ferdows und Barbour, 1989; Nosek et al., 1995; Wolff und Altenbuchner, 2000). Daher scheint die Hypothese, dass das erste lineare eukaryotische Chromosom (das von einem prokaryoten Vorfahren stammt) telomeraseunabhängig war, wahrscheinlicher., Es gibt einige Beispiele, die zeigen, dass das Telomerase-basierte System für die Telomerwartung bei allen Eukaryoten nicht wesentlich ist: Retrotransposons in Drosophila-Telomeren, Satellitenwiederholungen in Chironomus, einem anderen Insekt (Rubin, 1978; Biessmann und Mason, 2003) und ALT in Telomerase-negativen menschlichen Krebsarten (Hu et al., 2016; Zhang et al., 2019). Einige dieser Systeme sind jedoch möglicherweise nicht so unterschiedlich und haben möglicherweise einen gemeinsamen Ursprung: In Drosophila unterscheidet sich die Telomerase, die auf Retrotransposition basiert, nicht allzu sehr vom Telomerase-basierten Mechanismus (Danilevskaya et al.,, 1998), was zu der Hypothese führt, dass die Telomerase selbst ein ehemaliges Retrotransposon sein kann. Aber sicher, Telomerase-negative Pflanzenarten wurden bis heute nicht entdeckt und alle Ausnahmen, in denen das typische Pflanzentyp-Telomer fehlte, wurden später gezeigt, dass sie unterschiedliche, aber immer noch Telomerase-synthetisierte Motive haben. Dennoch ist die ALT-Maschinerie parallel zur Telomerase-Aktivität in Pflanzen vorhanden (Watson und Shippen, 2007; Ruckova et al., 2008). Daraus ergeben sich interessante Fragen zur Rolle von Telomerase, Telomeren und deren Aufrechterhaltung bei Pflanzentumoren., Ein attraktives Thema ist das Fehlen von Metastasen in Pflanzen, trotz des Vorhandenseins von ALT, möglicherweise im Zusammenhang mit der Starrheit des Pflanzengewebes oder einem anderen Immunsystem als bei Tieren (Seyfried und Huysentruyt, 2013).
Obwohl wir immer mehr Wissen über Telomerbiologie gewinnen, sind wir immer noch nicht in der Lage, die Entstehung von Telomerase in Eukaryoten zu erklären., Aktuelle Beweise stützen die Hypothese, dass die Entstehung von Eukaryoten zusammen mit ihren linearen Chromosomen, Telomeren und Telomerase mit dem Auftreten von spliceosomalen Intronen in archaealen Wirten zusammenhängt (Koonin, 2006; Fajkus et al., 2019). Die Ähnlichkeit zwischen TERT und anderen Retroelementen wird seit einiger Zeit diskutiert (Pardue et al., 1997). Bemerkenswert ist, dass eine relativ neue Studie zeigte, dass TERT als wahrscheinliches Mitglied der Nachkommen Gruppe II Introns, sequentiell in der Nähe Penelope-like Element retrotransposons (Gladyshev und Arkhipova, 2007)., TERT ist jedoch nur eine der beiden wesentlichen Telomerasekomponenten, und TR ist in seinem Ursprung aufgrund seiner geringen Sequenzkonservierung über alle Eukaryoten hinweg noch rätselhafter .
Schlussfolgerung
Zu Beginn der Pflanzengenomik galt die Telomersequenz als nahezu unveränderlich. Die allgemeine Erhaltung der Telomere und des Telomerasesystems deutete darauf hin, dass alle Pflanzen das TTTAGGG-Pflanzen-Telomer haben könnten. Die Identifizierung ungewöhnlicher Telomersequenzen in komplexen Pflanzengenomen, in vielen Fällen mit riesigen C-Werten (wie in Cestrum und Allium sp.,), war die Mühe Wert, da die außergewöhnlich lange Allium Telomer-Motiv war der Anhaltspunkt bei der Suche nach einer echten TR in Landpflanzen. Die neu beschriebene TR in Pflanzen und weitere Telomere/Telomerase-Forschung in basalen Algenklassen könnte wertvolle Informationen über die frühe Evolution aufdecken, Daher kann die Telomerforschung von Pflanzen signifikant zu Hypothesen über die Entstehung von Eukaryoten beitragen.
Autor Beiträge
VP und der SG beigetragen haben und ebenso für das schreiben, Bearbeiten und Vorbereitung dieses mini-review.,
Finanzierung
Interessenkonflikt
Die Autoren erklären, dass die Forschung ohne kommerzielle oder finanzielle Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.
Danksagung
Tausend Pflanze Transkriptoms Initiative (2019). Eintausend Pflanzentranskriptome und die Phylogenomik grüner Pflanzen. Natur 574 (7780), 679-685. doi: 10.1038 / s41586-019-1693-2
PubMed Abstract / CrossRef Volltext / Google Scholar
