Som du kan se i Figure1, den nukleotider bare variere litt, og bare i nitrogenholdige base. I tilfelle av DNA, disse basene er adenine, guanin, cytosine, og innhold av tymin. Merk likheten av de formene av adenine og guanin, og også likheten mellom cytosine og innhold av tymin. A og G er klassifisert som puriner, mens C og T er klassifisert som pyrimidines. Så lenge vi er navngi ting, legg merke til «deoxyribose» og «ribose». Som navnet tilsier, deoxyribose er bare en ribose uten oksygen., Mer spesifikt, hvor det er en hydroksyl gruppe knyttet til det 2-karbon av ribose, det er bare en hydrogen knyttet til det 2-karbon for deoxyribose. Det er den eneste forskjellen mellom de to sukker.
I tilfeldig konstruere et enkelt aspekt av nucleic acid in vitro, det er ingen særskilte regler om bestilling av nukleotider med hensyn til sine baser. Identiteten til sine nitrogenholdige baser er irrelevant fordi nukleotider er festet med phosphodiester obligasjoner gjennom fosfat gruppe og pentose., Det er derfor ofte referert til som sukker-fosfat ryggraden. Hvis vi bryter ned ordet «phosphodiester», ser vi at det er ganske praktisk beskriver sammenhengen: sukker er koblet sammen med to ester obligasjoner ( —O—) med en fosfor i mellom. En av ideene som ofte forvirrer studenter er det tekstretning av denne bindingen, og derfor, av nukleinsyrer generelt. For eksempel, når vi snakker om DNA-polymerase, enzymet som catalyzes tillegg av nukleotider i levende celler, sier vi at den fungerer på en 5-prime (5′) til å 3-prime (3′) retning., Dette kan virke som arcane molekylær-biolog-snakke, men det er faktisk veldig enkelt. Ta en titt på to av nukleotider sammen med den phosphodiester bond (Figur \(\PageIndex{1}\), nederst til venstre). En adenine nukleotid er koblet til et cytosine nukleotid. Den phosphodiester bond vil alltid knytte 5-karbon av en deoxyribose (eller ribose i RNA) til 3-karbon i neste sukker. Dette betyr også at på den ene enden av en kjede av koblede nukleotider, vil det være en fri 5′ – fosfat (-PO4) gruppe, og på den andre enden, en fri 3′ – hydroksyl (-OH)., Disse definere tekstretning av en strand av DNA eller RNA.

DNA er vanligvis finnes som en dobbel-strandet molekyler i cellen, mens RNA er for det meste enkelt-strandet., Det er viktig å forstå om, at under egnede forhold, DNA kan gjøres enkelt-strandet, og RNA kan være dobbel-strandet. Faktisk, molekylene er så like at det er enda mulig å lage dobbel-strandet hybrid molekyler med en tråd av DNA og en av RNA. Interessant, RNA-RNA dobbeltrom helices og RNA-DNA-dobbel helices er faktisk litt mer stabil enn den mer konvensjonelle DNA-DNA-dobbel helix.,
bakgrunn av dobbel-strandet arten av DNA, og faktisk grunnlag av nukleinsyrer som medium for lagring og overføring av genetisk informasjon, er base-sammenkobling. Base-sammenkobling refererer til dannelse av hydrogen bindinger mellom adenines og thymines, og mellom guanines og cytosines. Disse parene er betydelig mer stabil enn noen forening dannet med andre mulige baser. Videre, når disse base-par foreninger form i sammenheng med to tråder av nukleinsyrer, deres avstand er også uniform og stabil., Du husker kanskje at hydrogen obligasjoner er relativt svake bånd. Imidlertid, i forbindelse med DNA, hydrogenbinding er det som gjør DNA-ekstremt stabile og derfor godt egnet som en langsiktig lagringsmedium for genetisk informasjon. Siden selv i enkle prokaryotes, DNA dobbeltrom helices er minst tusenvis av nukleotider lange, betyr dette at det er flere tusen hydrogen obligasjoner holde de to landene sammen., Selv om noen enkelt nukleotid-til-nukleotid hydrogenbinding samhandling lett kunne bli midlertidig forstyrret av en svak økning i temperatur, eller en minimale endringer i den joniske styrke av løsningen, en full dobbel-helix DNA krever svært høye temperaturer (vanligvis over 90oC) til helt denaturering dobbel helix i individuelle tråder.
Fordi det er en presis en-til-en sammenkobling av nukleotider, viser det seg at de to trådene er i hovedsak backup kopier av hverandre – et sikkerhetsnett i tilfelle at nukleotider er mistet fra en strand., Faktisk, selv om deler av begge trådene er skadet, så lenge den andre strand er intakt i området av skade, så essensiell informasjon er fortsatt det i de utfyllende sekvens av det motsatte strand og kan skrives på plass. Husk skjønt, at mens en strand av DNA kan dermed fungere som en «backup» av de andre, de to trådene er ikke identiske – de er komplementære. En interessant konsekvens av dette systemet av komplementær og antiparallel tilnærmingene er at de to tilnærmingene kan hver bære unik informasjon.,
Bi-directional gen-par er to gener som er på motsatt side av trådene i DNA, men deling av arrangøren, som ligger i mellom dem. Siden DNA kan bare gjøres i én retning, 5′ til 3′, dette bi-directional arrangøren, ofte en CpG island (se neste kapittel), og dermed sender RNA polymerase for hvert gen i motsatt fysiske retninger. Dette har blitt vist for en rekke gener som er involvert i kreft (bryst, eggstokk), og er en mekanisme for å koordinere uttrykk av et nettverk av genet produkter.
trådene i DNA-dobbel-helix er antiparallel., Dette betyr at hvis vi så på en dobbel-helix DNA fra venstre til høyre, en strand ville bli bygget i 5′ til 3′ retning, mens den komplementære tråden er bygget i 3′ til 5′ retning. Dette er viktig for funksjonen av enzymer som kan skape og reparasjon av DNA, som vi skal drøfte snart. I Figur \(\PageIndex{1}\), venstre strand er 5′ til 3′ fra topp til bunn, og den andre er 5′ til 3′ fra bunn til topp.
Fra et fysisk synspunkt, DNA-molekyler som er negativt ladet (alle de fosfater), og normalt er det en dobbel-helix med en høyrehendt vri., I denne normal (også kalt «B» konformasjon) staten, en full vri av molekylet omfatter 11 base-par, med 0.34 nm mellom hvert nukleotid base. Hver av nitrogenholdige baser er plane, og da sammen med utfyllende base, danner en på plane «trinn» på «stigen» av DNA. Disse er vinkelrett på lengdeaksen av DNA. De fleste av fritt-flytende DNA i en celle, og de fleste DNA i alle vandig løsning av nær-fysiologiske osmolaritet og pH, er funnet i denne B konformasjon., Imidlertid, andre conformations har blitt funnet, vanligvis under svært spesielle miljøforhold. En komprimert konformasjon, EN-DNA, ble observert som en konsekvens av in vitro-krystallisering, med litt mer baser per tur, kortere slå lengde, og base-par som er vinkelrett på lengdeaksen. En annen, Z-DNA, ser ut til å danne kortvarig i GC-rike strekninger av DNA-et som, interessant, DNA vendinger motsatt retning.
Det har blitt foreslått at både A og Z former for DNA er, faktisk, fysiologisk relevante., Det er bevis som tyder på at En form kan oppstå i RNA-DNA-hybrid dobbeltrom helices samt når DNA er complexed til noen enzymer. Z-konformasjon kan oppstå i respons til metylering av DNA. Videre er «normal» B-DNA konformasjon er noe av en idealisert struktur basert på å være fullt hydrert, som er helt sikkert svært sannsynlig inne i en celle. Men, som fuktighets-stat, er i stadig endring, men grundig, slik at DNA-konformasjon vil ofte variere litt fra B-konformasjon parametrene i Figur \(\PageIndex{2}\).,
I prokaryotes, DNA er funnet i cytoplasma (ganske opplagt, siden det er ingen andre valg i disse enkle organismer), mens i eukaryotes, DNA finnes i cellekjernen. Til tross for forskjellene i deres steder, nivået av beskyttelse fra eksterne krefter, og mest av alt, deres størrelser, både prokaryotic og eukaryote DNA er pakket med proteiner som hjelper til å organisere og til å stabilisere den samlede kromosom struktur., Relativt lite er forstått med hensyn til prokaryotic kromosomale emballasje selv om det er strukturelle likheter mellom noen av proteinene som finnes i prokaryotic og eukaryote kromosomer. Derfor, de fleste innledende cellebiologi kurs hold deg til eukaryote kromosomale emballasje.

Naken DNA, enten prokaryotic eller eukaryote, er en ekstremt tynn tråd av materiale, med om lag 11 nm i diameter. Imidlertid, gitt størrelsen av eukaryote genomer, hvis DNA ble lagret på den måten inne i kjernen, det ville bli unmanageably tangled. Bilde en bøtte som du har kastet en hundre meter med garn uten noen forsøk overhodet å organisere det ved coiling det eller bunching det., Nå må du vurdere om du ville være i stand til å nå inn som spann dra på en strand, og forventer å trekke opp bare en strand, eller hvis stedet du er sannsynlig å trekke opp minst en liten floke av garn. Cellen i hovedsak gjør hva du vil gjøre med garn for å holde den i orden: det er pakket pent inn i mindre, håndterbare skeins. I tilfelle av DNA, hvert kromosom er en løkke rundt en histone komplisert å danne den første bestillingen av kromosomavvik organisasjon: nucleosome.
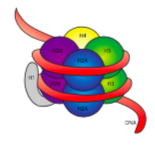
Den 30-nm fiber er holdt sammen av to sett av interaksjoner. Først, den som lenker histone, H1, bringer nucleosomes sammen til en omtrentlig 30-nm struktur., Denne strukturen er så stabiliseres av disulfide obligasjoner som dannes mellom H2A histone av en nucleosome og H4 histone av sin nabo.
Histones er en familie på grunnleggende (positivt ladet) proteiner. De fungerer først og fremst i å organisere DNA, og nucleosome er dannet når DNA-wraps (litt over 2 ganger) rundt en kjerne av åtte histones – to hver av H2A, H2B, H3 og H4. Antall og plassering av positive ladninger (for det meste fra lysines og arginines) er avgjørende for deres evne til tett binder DNA, som tidligere påpekt, er svært negativt ladet., Som «motsetninger tiltrekker hverandre» ideen er ikke bare en dating tips fra råd kolonner.

Ved undersøkelse av 3D struktur av histone core komplisert, ser vi at mens det var relativt ladet protein samhandling domener hold histones sammen i midten, den positivt ladede rester er funnet rundt utsiden av komplekset, som er tilgjengelig til å samhandle med negativt ladet fosfater av DNA.,
I et senere kapittel, vil vi diskutere hvordan enzymer lese DNA for å transkribere sin informasjon på mindre, mer håndterbare biter av RNA. For nå, vi trenger bare å være klar over at til enhver tid, er mye av DNA er pakket tett unna, mens noen deler av DNA-et som ikke er det. Fordi de deler som er tilgjengelig for bruk kan variere avhengig av hva som skjer til/i cellen til enhver tid, pakking av DNA må være dynamisk. Det må være en mekanisme for å raskt løsne binding av DNA til histones når det DNA som er nødvendig for genuttrykk, og å stramme bindende når det er det ikke., Som det viser seg, kan denne prosessen innebærer acetylation og deacetylation av histones.

Histone Acetyltransferases (Hatter) er enzymer som plasserer en acetyl-gruppen på en lysin av en histone protein. Den acetyl grupper er negativt ladet, og acetylation ikke bare legger et negativt ladet gruppe, det fjerner også den positive ladningen fra lysin. Dette har effekten av ikke bare å nøytralisere et punkt om tiltrekning mellom protein og DNA, men enda litt frastøtende det (med like ladninger)., På den andre siden av mekanismen, Histone Deactylases (HDACs) er enzymer som fjerner acetylation, og dermed gjenopprette samspillet mellom histone protein og DNA. Siden disse er så viktige enzymer, det står til grunn at de ikke får lov til å operere willy-bulle på hvilken som helst tilgjengelig histone, og faktisk, de er ofte funnet i et kompleks med andre proteiner som kontrollerer og koordinerer sine aktivering med andre prosesser, som for eksempel aktivering av transkripsjon.
