como você pode ver na Figura 1, os nucleótidos apenas variam ligeiramente, e apenas na base azotada. No caso do DNA, essas bases são adenina, guanina, citosina e timina. Note a similaridade das formas de adenina e guanina, e também a similaridade entre citosina e timina. A E G são classificadas como purinas, enquanto C E T são classificadas como pirimidinas. Já que estamos a dar nomes às coisas, notem “desoxirribose” e “ribose”. Como o nome indica, a desoxirribose é apenas uma ribose sem oxigénio., Mais especificamente, onde há um grupo hidroxilo ligado ao 2-carbono da ribose, há apenas um hidrogênio ligado ao 2-carbono da desoxirribose. Essa é a única diferença entre os dois açúcares.
na construção aleatória de uma única cadeia de ácido nucleico in vitro, não existem regras particulares relativas à ordenação dos nucleótidos em relação às suas bases. As identidades de suas bases azotadas são irrelevantes porque os nucleótidos são ligados por ligações fosfodiésteres através do grupo fosfato e da pentose., É, portanto, muitas vezes referido como a espinha dorsal do açúcar-fosfato. Se quebrarmos a palavra “fosfodiéster”, vemos que ela descreve muito habilmente a conexão: os açúcares são conectados por duas ligações ester ( —O—) com um fósforo no meio. Uma das ideias que muitas vezes confunde estudantes é a direcionalidade desta ligação, e portanto, dos ácidos nucleicos em geral. Por exemplo, quando falamos de DNA polimerase, a enzima que catalisa a adição de nucleótidos em células vivas, dizemos que ela funciona em uma direção de 5-prime (5′) a 3-prime (3′)., Isto pode parecer Arcano molecular-biólogo-falar, mas na verdade é muito simples. Dê outra olhada em dois dos nucleótidos Unidos pela ligação fosfodiéster (figura \(\PageIndex{1}\), inferior esquerda). Um nucleótido de adenina é unido a um nucleótido de citosina. A ligação fosfodiéster sempre ligará o 5-carbono de uma desoxirribose (ou ribose no RNA) ao 3-carbono do próximo açúcar. Isto também significa que em uma extremidade de uma cadeia de nucleótidos ligados, haverá um grupo 5′ fosfato livre (-PO4), e na outra extremidade, um 3′ hidroxilo livre (-OH)., Estes definem a direcionalidade de uma cadeia de DNA ou RNA.

DNA é normalmente encontrado como uma molécula de cadeia dupla na célula, enquanto o RNA é principalmente de cadeia simples., É importante entender, porém, que sob as condições apropriadas, o DNA pode ser feito de cadeia única, e o RNA pode ser de cadeia dupla. De fato, as moléculas são tão semelhantes que é possível até mesmo criar moléculas híbridas de cadeia dupla com uma cadeia de DNA e uma de RNA. Curiosamente, as hélices duplas RNA-RNA e RNA-DNA são ligeiramente mais estáveis do que a hélice dupla DNA-DNA mais convencional.,
a base da natureza de cadeia dupla do ADN e, na verdade, a base dos ácidos nucleicos como meio de armazenamento e transferência de informação genética, é o emparelhamento de base. Emparelhamento de Base refere-se à formação de ligações de hidrogênio entre adeninas e timinas, e entre guaninas e citosina. Estes pares são significativamente mais estáveis do que qualquer associação formada com as outras bases possíveis. Além disso, quando estas associações de pares de bases se formam no contexto de duas cadeias de ácidos nucleicos, o seu espaçamento também é uniforme e altamente estável., Devem lembrar-se que as ligações de hidrogénio são ligações relativamente fracas. No entanto, no contexto do ADN, a ligação do hidrogénio é o que torna o ADN extremamente estável e, portanto, adequado como meio de armazenamento a longo prazo para a informação genética. Uma vez que mesmo em procariontes simples, hélices duplas de DNA são pelo menos milhares de nucleotídeos de comprimento, isso significa que existem vários milhares de ligações de hidrogênio segurando as duas cadeias juntas., Embora qualquer indivíduo de nucleotídeos-para-nucleotídeo hidrogénio vínculo de interação poderia facilmente ser temporariamente interrompido por um ligeiro aumento de temperatura, ou uma minúscula alteração na força iônica da solução, uma dupla-hélice do DNA requer temperaturas muito altas (geralmente mais de 90oC) completamente desnaturar a dupla hélice para fios individuais.
porque há uma combinação exata de nucleótidos, acontece que as duas cadeias são essencialmente cópias de backup uma da outra-uma rede de segurança no caso de os nucleótidos serem perdidos de uma cadeia., Na verdade, mesmo que partes de ambos os cordões estejam danificadas, desde que o outro filete esteja intacto na área de danos, então a informação essencial ainda está lá na sequência complementar do filamento oposto e pode ser escrito no lugar. Tenha em mente, porém, que enquanto uma cadeia de DNA pode assim agir como um “backup” da outra, as duas cadeias não são idênticas – elas são complementares. Uma consequência interessante deste sistema de vertentes complementares e antiparalelas é que cada uma das duas vertentes pode ter informações únicas.,os pares de genes bidireccionais
são dois genes em cadeias opostas de ADN, mas partilham um promotor, que se encontra entre eles. Uma vez que o DNA só pode ser feito em uma direção, 5′ a 3′, Este promotor bidirecional, muitas vezes uma ilha CpG (ver capítulo seguinte), assim envia a RNA polimerase para cada gene em direções físicas opostas. Isto tem sido demonstrado para uma série de genes envolvidos em cancros (mama, ovário), e é um mecanismo para coordenar a expressão de redes de produtos genéticos.
as cadeias de uma dupla hélice de ADN são antiparalelas., Isto significa que se olharmos para uma dupla hélice de DNA da esquerda para a direita, uma cadeia seria construída na direção 5′ a 3′, enquanto a cadeia complementar é construída na direção 3′ a 5′. Isto é importante para a função das enzimas que criam e reparam o ADN, como iremos discutir em breve. Na figura \(\PageIndex{1}\), a linha esquerda é de 5′ a 3′ de cima para baixo, e a outra é de 5′ a 3′ de baixo para cima.
do ponto de vista físico, moléculas de DNA são carregadas negativamente (todos esses fosfatos), e normalmente uma dupla hélice com uma torção destraada., Neste estado normal (também chamado de conformação “B”), um torção completo da molécula engloba 11 pares de base, com 0,34 nm entre cada base nucleotídica. Cada uma das bases azotadas são planar, e quando emparelhada com a base complementar, forma um “degrau” em planar na “escada” do DNA. Estes são perpendiculares ao eixo longitudinal do DNA. A maioria do DNA flutuante livre em uma célula, e a maioria do DNA em qualquer solução aquosa de osmolaridade e pH quase fisiológicos, é encontrada nesta conformação B., No entanto, foram encontradas outras conformações, geralmente em circunstâncias ambientais muito específicas. Uma conformação comprimida, a-DNA, foi observada como um artefato de cristalização in vitro, com um pouco mais de bases por volta, menor comprimento de volta, e pares de bases que não são perpendiculares ao eixo longitudinal. Outro, o DNA-Z, parece formar-se transitoriamente em extensões ricas em GC de DNA em que, curiosamente, o DNA distorce a direção oposta.
tem sido sugerido que ambas as formas A E Z de ADN são, de facto, fisiologicamente relevantes., Existem evidências que sugerem que a forma A pode ocorrer em hélices duplas híbridas RNA-DNA, bem como quando o DNA está complexado a algumas enzimas. A conformação Z pode ocorrer em resposta à metilação do ADN. Além disso, a conformação” normal ” do DNA B é algo de uma estrutura idealizada baseada em ser totalmente hidratada, como é certamente muito provável dentro de uma célula. No entanto, esse estado de hidratação está constantemente mudando, ainda que minuciosamente, de modo que a conformação do DNA muitas vezes varia ligeiramente a partir dos parâmetros de conformação B Na Figura \(\PageIndex{2}\).,
em procariontes, o DNA é encontrado no citoplasma (bastante óbvio, uma vez que não há outra escolha nesses organismos simples), enquanto que em eucariontes, o DNA é encontrado dentro do núcleo. Apesar das diferenças em suas localizações, o nível de proteção de forças externas, e, acima de tudo, seus tamanhos, tanto em procariontes e eucarióticas DNA é embalado com proteínas que ajudam a organizar e estabilizar geral da estrutura dos cromossomas., Relativamente pouco se entende em relação à embalagem cromossômica procariótica, embora haja semelhanças estruturais entre algumas das proteínas encontradas nos cromossomos procarióticos e eucarióticos. Por conseguinte, a maioria dos cursos de biologia celular introdutórios mantém-se na embalagem cromossómica eucariótica.

DNA Nu, quer seja procariótico ou eucariótico, é uma cadeia extremamente fina de material, com aproximadamente 11 nm de diâmetro. No entanto, dado o tamanho dos genomas eucarióticos, se o ADN fosse armazenado dessa forma dentro do núcleo, tornar-se-ia incontrolável. Imagine um balde em que você jogou uma centena de metros de fio, sem qualquer tentativa de organizá-lo por enrolá-lo ou bunching-lo., Agora considere se você seria capaz de chegar a esse balde puxar em um fio, e esperar para puxar apenas um fio, ou se em vez disso você é susceptível de puxar para cima pelo menos um pequeno emaranhado de fios. A célula faz essencialmente o que você faria com o fio para mantê-lo organizado: ele é embalado perfeitamente em pequenos, skeins gerenciáveis. No caso do DNA, cada cromossomo é looped em torno de um complexo histone para formar a primeira ordem da organização cromossômica: o nucleossomo.
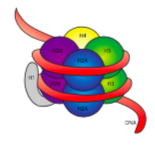
a fibra de 30 nm é mantida unida por dois conjuntos de interações. Primeiro, o linker histone, H1, reúne os nucleossomas em uma estrutura de aproximadamente 30 nm., Esta estrutura é então estabilizada por ligações dissulfeto que se formam entre a histona H2A de um nucleossomo e a histona H4 de seu vizinho.histonas são uma família de proteínas básicas (positivamente carregadas). Todos eles funcionam principalmente na organização do DNA, e o nucleossomo é formado quando o DNA envolve (um pouco mais de 2 vezes) em torno de um núcleo de oito histonas – duas cada uma de H2A, H2B, H3, e H4. O número e a posição das cargas positivas (principalmente de lisinas e argininas) são cruciais para a sua capacidade de se ligarem firmemente ao DNA, que como já foi apontado, é muito negativamente carregado., Essa ideia de “opostos atraem” não é apenas uma dica das colunas de conselhos.

Após o exame da estrutura 3D da histona núcleo complexo, vemos que apesar de ser relativamente vazia de proteína de interação domínios mantenha as histonas juntos no centro, a carga positiva resíduos são encontrados do lado de fora do complexo, disponível para interagir com a carga negativa fosfatos do DNA.,
em um capítulo posterior, discutiremos como as enzimas lêem o DNA para transcrever sua informação em pedaços menores e mais gerenciáveis de RNA. Por agora, só precisamos estar cientes de que, a qualquer momento, grande parte do DNA é embalado firmemente longe, enquanto algumas partes do DNA não são. Porque as partes que estão disponíveis para uso podem variar dependendo do que está acontecendo com/na célula em qualquer momento, a embalagem de DNA deve ser dinâmica. Deve haver um mecanismo para soltar rapidamente a ligação do DNA às histonas quando esse DNA é necessário para a expressão do gene, e para apertar a ligação quando não é., Ao que parece, este processo envolve acetilação e desacetilação das histonas.

Acetiltransferases histona (chapéus) são enzimas que colocam um grupo acetilo numa lisina de uma proteína da histona. Os grupos acetilo são negativamente carregados, e a acetilação não só adiciona um grupo negativamente carregado, ele também remove a carga positiva da lisina. Isto tem o efeito de não só neutralizar um ponto de atração entre a proteína e o DNA, mas até mesmo repeli-lo ligeiramente (com cargas semelhantes)., Do outro lado do mecanismo, Histone Deactylases (HDACs) são enzimas que removem a acetilação, restabelecendo assim a interacção entre a proteína histona e o ADN. Uma vez que estes são tão importantes enzimas, é lógico que eles não estão autorizados a operar voluntariamente ou não em qualquer histona, e na verdade, eles são freqüentemente encontrados em um complexo com outras proteínas que controlam e coordenam a sua ativação com outros processos, tais como a ativação da transcrição.
